Crédito: Protocolos de la naturaleza (2024). DOI: 10.1038/s41596-024-01046-3
Un equipo de investigación dirigido por científicos de la Universidad de California en Riverside ha desarrollado un flujo de trabajo computacional para analizar grandes conjuntos de datos en el campo de la metabolómica, el estudio de pequeñas moléculas que se encuentran dentro de células, biofluidos, tejidos y ecosistemas enteros.
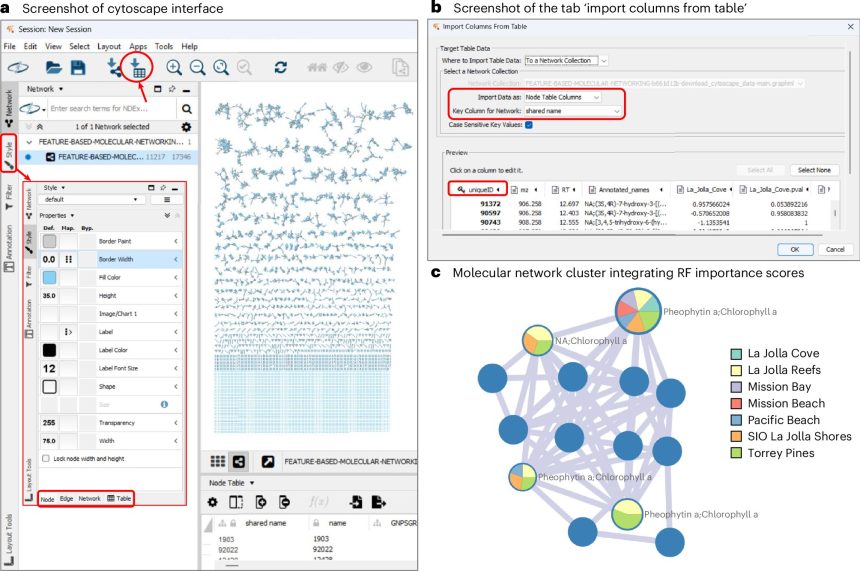
Recientemente, el equipo aplicó esta nueva herramienta computacional para analizar los contaminantes del agua de mar en el sur de California. El equipo capturó rápidamente los perfiles químicos de los entornos costeros y destacó las posibles fuentes de contaminación.
«Nos interesa comprender cómo se introducen estos contaminantes en el ecosistema», dijo Daniel Petras, profesor adjunto de bioquímica en la UC Riverside, quien dirigió el equipo de investigación. «Determinar qué moléculas del océano son importantes para la salud ambiental no es sencillo debido a la gran diversidad química del océano. El protocolo que desarrollamos acelera enormemente este proceso. Una clasificación más eficiente de los datos significa que podemos comprender los problemas relacionados con la contaminación del océano más rápidamente».
Petras y sus colegas informe En el diario Protocolos de la naturaleza Su protocolo está diseñado no solo para investigadores experimentados, sino también con fines educativos, lo que lo convierte en un recurso ideal para estudiantes y científicos que recién comienzan su carrera. Este flujo de trabajo computacional está acompañado de una aplicación web accesible con una interfaz gráfica de usuario que hace que el análisis de datos metabolómicos sea accesible para los no expertos y les permite obtener información estadística de sus datos en cuestión de minutos.
«Esta herramienta es accesible para una amplia gama de investigadores, desde principiantes absolutos hasta expertos, y está diseñada para usarse junto con el software de redes moleculares que mi grupo está desarrollando», dijo el coautor Mingxun Wang, profesor adjunto de ciencias de la computación e ingeniería en la UCR. «Para los principiantes, las pautas y el código que proporcionamos facilitan la comprensión de los pasos comunes de procesamiento y análisis de datos. Para los expertos, acelera el análisis de datos reproducibles, lo que les permite compartir sus flujos de trabajo y resultados de análisis de datos estadísticos».
Petras explicó que el artículo de investigación es único y que sirve como un gran recurso educativo organizado a través de un grupo de investigación virtual llamado Virtual Multiomics Lab o VMOL. Con más de 50 científicos participantes de todo el mundo, VMOL es una comunidad de acceso abierto impulsada por la comunidad. Su objetivo es simplificar y democratizar el proceso de análisis químico, haciéndolo accesible a investigadores de todo el mundo, independientemente de su formación o recursos.
«Estoy increíblemente orgulloso de ver cómo este proyecto se convirtió en algo impactante, en el que participaron expertos y estudiantes de todo el mundo», dijo Abzer Pakkir Shah, estudiante de doctorado del grupo de Petras y primer autor del artículo. «Al eliminar las barreras físicas y económicas, VMOL ofrece capacitación en espectrometría de masas computacional y ciencia de datos y tiene como objetivo lanzar proyectos de investigación virtuales como una nueva forma de ciencia colaborativa».
Todo el software desarrollado por el equipo es gratuito y está disponible públicamente. El desarrollo del software se inició durante una escuela de verano sobre metabolómica no dirigida en 2022 en la Universidad de Tubinga, donde el equipo también lanzó VMOL.
Petras espera que el protocolo sea especialmente útil para los investigadores ambientales, así como para los científicos que trabajan en el campo biomédico y los investigadores que realizan estudios clínicos en la ciencia del microbioma.
«La versatilidad de nuestro protocolo se extiende a una amplia gama de campos y tipos de muestras, incluida la química combinatoria, el análisis de dopaje y la contaminación por trazas de alimentos, productos farmacéuticos y otros productos industriales», dijo.
Petras obtuvo su maestría en biotecnología en la Universidad de Ciencias Aplicadas de Darmstadt y su doctorado en bioquímica en la Universidad Técnica de Berlín. Realizó una investigación posdoctoral en la UC San Diego, donde se centró en el desarrollo de métodos de metabolómica ambiental a gran escala. En 2021, puso en marcha el Laboratorio de Metabolómica Funcional en la Universidad de Tübingen. En enero de 2024 se incorporó a la UCR, donde su laboratorio se centra en el desarrollo y la aplicación de métodos basados en espectrometría de masas para visualizar y evaluar el intercambio químico dentro de las comunidades microbianas.
Más información:
Abzer K. Pakkir Shah et al, Análisis estadístico de los resultados de redes moleculares basadas en características a partir de datos metabolómicos no específicos, Protocolos de la naturaleza (2024). DOI: 10.1038/s41596-024-01046-3
Citación:Una nueva herramienta de ciencia de datos acelera enormemente el análisis molecular de nuestro entorno (20 de septiembre de 2024) recuperado el 21 de septiembre de 2024 de https://phys.org/news/2024-09-science-tool-greatly-molecular-analysis.html
Este documento está sujeto a derechos de autor. Salvo que se haga un uso legítimo con fines de estudio o investigación privados, no se podrá reproducir ninguna parte del mismo sin autorización por escrito. El contenido se ofrece únicamente con fines informativos.