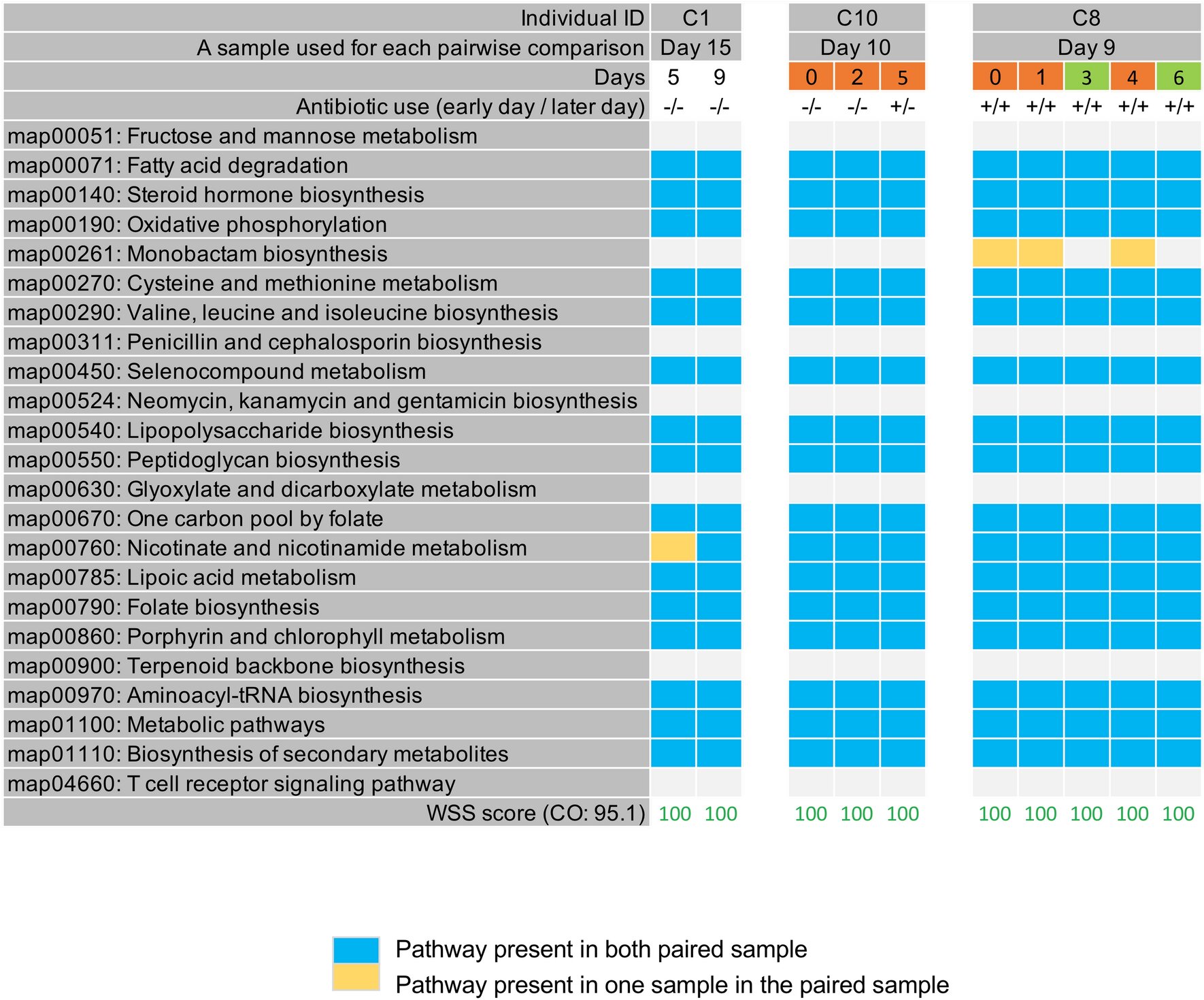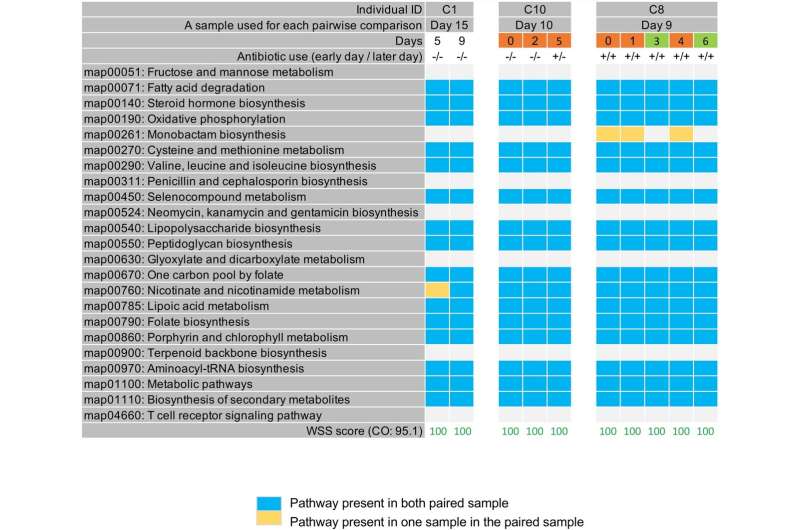
PKS resulta de personas hospitalizadas con COVID-19. Se utilizó un total de 23 vías KEGG para examinar un patrón de presencia/ausencia de vías metabólicas KEGG para B. vulgatus y se observó la presencia o ausencia de cada vía KEGG comparando la muestra del último día de cada paciente con cada par posible de muestras del mismo paciente. muestras Todas las muestras de pacientes fueron recolectadas previamente por Zuo et al. 17. El resultado PKS compartido por paciente se agrupó en cuadros de diferentes colores. Cada columna de la tabla indica el ID individual, una muestra utilizada para cada comparación por pares y los días. Crédito: Informes científicos (2022). DOI: 10.1038/s41598-022-10472-w
Casey D. Morrow, Ph.D., y sus colegas de la Universidad de Alabama en Birmingham desarrollaron previamente un método de «huella digital» de microbioma llamado WSS que identifica cepas individuales de bacterias intestinales particulares, a través del análisis de datos metagenómicos de muestras fecales. Han demostrado que cepas particulares en adultos tienden a permanecer estables con el tiempo, a menos que sean perturbadas por eventos como antibióticos o cirugía para la obesidad. También vieron que una cepa de trasplante fecal de donante administrada para tratar infecciones por Clostridium difficile resistente a los medicamentos persistió en el receptor hasta dos años después del trasplante.
Morrow y Hyunmin Koo, Ph.D., refinaron el método de huellas dactilares para incluir la búsqueda de variantes de un solo nucleótido en las vías metabólicas de KEGG de una cepa en particular. Estas variantes pueden identificar subcepas de una sola cepa identificada por WSS. Para observar las subcepas de una cepa de Bacteroides vulgatus, por ejemplo, Morrow y Koo examinaron 23 vías metabólicas KEGG diferentes presentes en esa bacteria.
Ahora han aplicado este análisis ampliado para monitorear los cambios en las subcepas durante períodos de tiempo más cortos, días o semanas, en dos bacterias intestinales clave: B. vulgatus y Bacteroides uniformis. Comparando un pequeño número de individuos sanos y pacientes hospitalizados con COVID-19, ven una diferencia en la dinámica de la subtensión que, según dicen, presagia una desaceleración de las tasas intrínsecas de variación de la tensión en los pacientes enfermos. Esta desaceleración podría conducir eventualmente a una disbiosis en la comunidad de cepas microbianas que podría presagiar un cambio en las cepas dominantes del microbioma intestinal.
Ambas especies de Bacteroides se encuentran en gran abundancia en la flora intestinal y pueden ser especies clave, organismos que ayudan a definir un ecosistema completo.
El estudio de Koo y Morrow, «Indicadores tempranos de disbiosis de cepa microbiana en la comunidad microbiana gastrointestinal humana de ciertos humanos sanos y pacientes hospitalizados con COVID‑19», se publica en la revista Informes científicos.
Koo y Morrow primero analizaron datos metagenómicos publicados previamente de 41 individuos muestreados con un año de diferencia y 11 individuos muestreados con 90 días de diferencia. Examinaron una sola cepa dominante de B. vulgatus en cada individuo en los dos momentos para ver si habían mostrado diferentes patrones de subcepas metabólicas de KEGG, como se detectó a partir del análisis de variantes de un solo nucleótido en las vías metabólicas de KEGG, o PKS. En general, la mayoría mostró un patrón de PKS de subcepa diferente entre los dos puntos de tiempo de cada individuo.
Luego, los investigadores de la UAB analizaron datos metagenómicos publicados previamente de seis individuos sanos muestreados cada pocos días durante tres a 10 semanas, analizando nuevamente subcepas por variantes de un solo nucleótido en 23 vías metabólicas KEGG. Tres individuos mostraron una subcepa diferente en cada punto de tiempo, mientras que tres subcepas mostraron patrones de PKS que aparecían, desaparecían y reaparecían en diferentes puntos de tiempo.
También se observaron patrones de PKS compartidos en dos de los tres pacientes hospitalizados con COVID-19 que fueron muestreados varias veces.
«Sugerimos que las comunidades microbianas intestinales bajo estrés, como las que se encuentran en pacientes hospitalizados con COVID-19, podrían estar en un estado que indica el cambio potencial en el que la cepa dominante sería superada por una cepa menor», dijo Koo. «Las interrupciones de la comunidad microbiana intestinal resultantes de una variación de cepa podrían, a su vez, alterar la estructura de la comunidad e impactar las funciones en el metabolismo y la resistencia a la colonización».
«Una de las características de un sistema biológico complejo es que, a medida que se acerca a una transición crítica, hay una desaceleración de las tasas de cambio intrínsecas», dijo Morrow. «El sistema entra en una condición relacionada con la autocorrelación, donde los patrones se repetirían entre puntos de tiempo. Es posible que los grupos de vías metabólicas KEGG compartidas representen un estado de autocorrelación en la comunidad de cepas microbianas intestinales que presagia un cambio de cepa».
Las cepas microbianas muestran patrones individualizados de estabilidad en el intestino infantil en desarrollo
Hyunmin Koo et al, Indicadores tempranos de disbiosis de cepa microbiana en la comunidad microbiana gastrointestinal humana de ciertos humanos sanos y pacientes hospitalizados con COVID-19, Informes científicos (2022). DOI: 10.1038/s41598-022-10472-w
Citación: Un método de ‘huella digital’ de microbioma refinado rastrea variantes de una sola cepa de microbio intestinal (27 de abril de 2022) recuperado el 27 de abril de 2022 de https://medicalxpress.com/news/2022-04-refined-microbiome-fingerprint-method-tracks .html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.