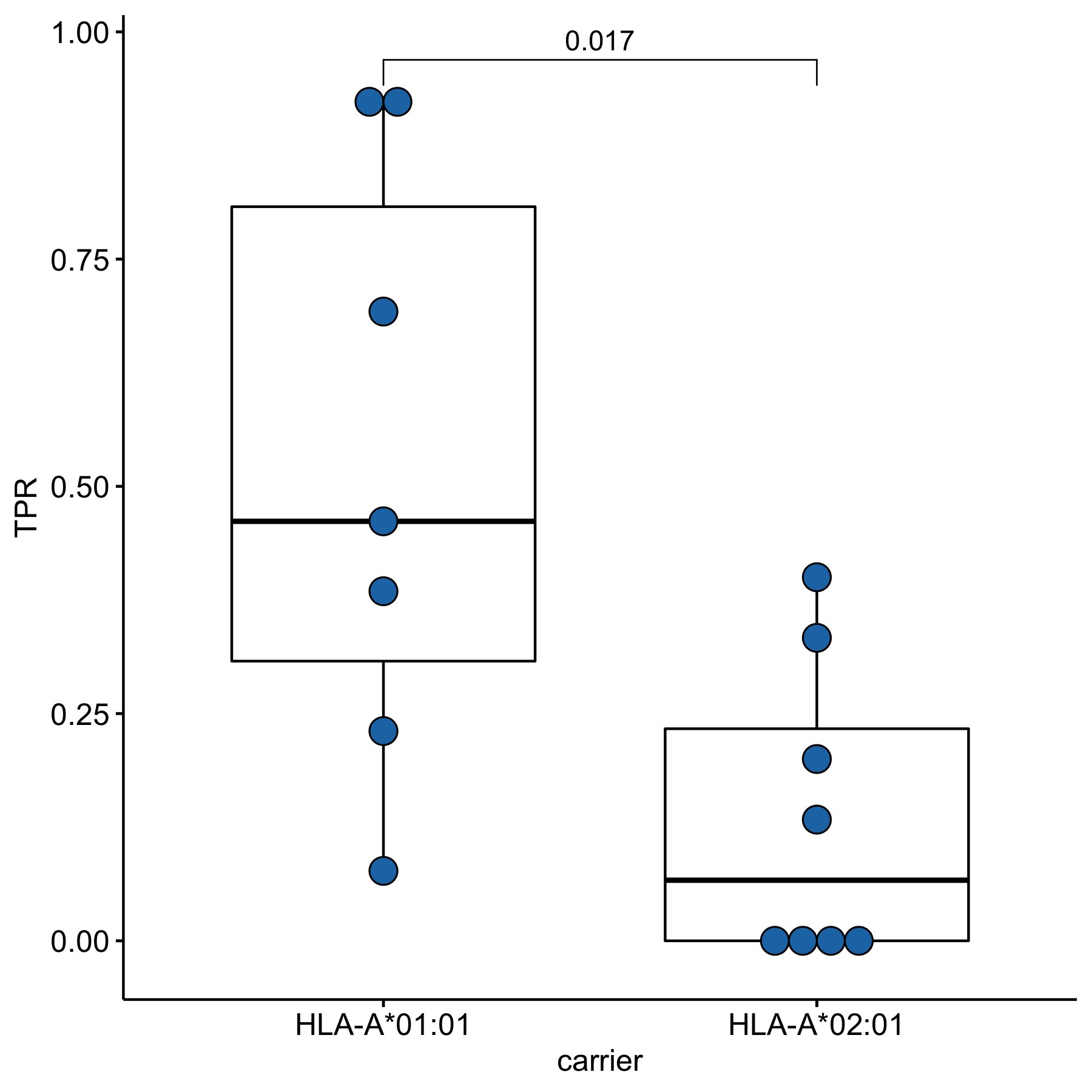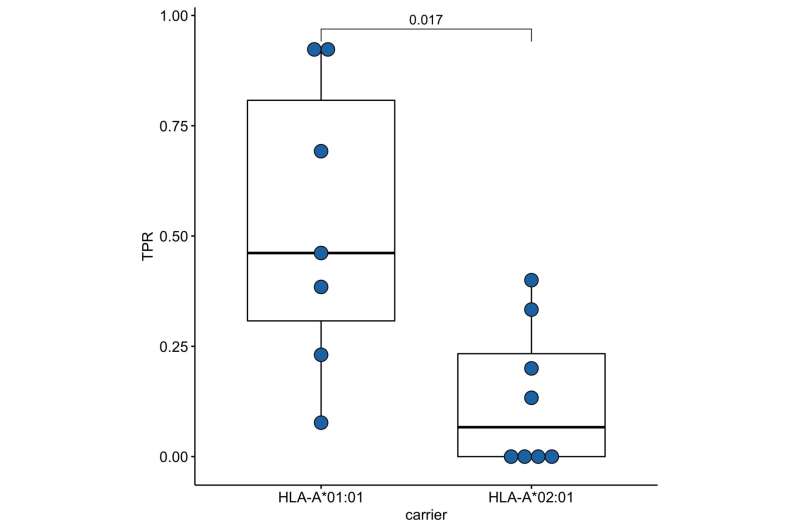
Validación de la inmunogenicidad de los epítopos ORF1ab en un subconjunto de portadores HLA-A*01:01 y HLA-A*02:01 convalecientes. Tasas positivas verdaderas para el epítopo ORF1ab en los grupos de comparación. Crédito: 2023 Shkurnikov et al.
La variante delta del SARS-CoV-2 que provocó la tercera ola de COVID-19 a mediados de 2021 resultó ser más contagiosa que las variantes anteriores del SARS-CoV-2. Además, se descubrió que las mutaciones de proteínas en la variante delta reducen significativamente el efecto de la inmunidad humoral adquirida a COVID-19 de una infección o vacunación previa.
La inmunidad adquirida a las infecciones la proporcionan en gran medida los linfocitos T, células del sistema inmunitario que detectan antígenos extraños después de la infección y desencadenan una respuesta inmunitaria. Este es el papel de los receptores de células T (o TCR), receptores especiales en la superficie de las células T.
Los TCR son responsables del reconocimiento de patógenos. Pueden «recordar» antígenos específicos encontrados previamente y montar una respuesta inmune más rápido la próxima vez, protegiendo así al cuerpo contra la reinfección con el mismo virus.
Las moléculas de antígeno leucocitario humano clase I (HLA-I) ayudan a los TCR a reconocer patógenos. Las moléculas HLA-I se unen a las moléculas del patógeno y las presentan en la superficie de la célula infectada, donde pueden ser reconocidas por los receptores de células T.
El conjunto de genes HLA-I de un individuo es único. Esta es la razón principal por la que las infecciones virales afectan a las personas con diversos grados de gravedad. En particular, como los investigadores de HSE demostraron anteriormente, el genotipo HLA-I individual determina la predisposición a formas graves de COVID-19.
Sin embargo, hasta ahora, los genotipos de los pacientes solo se estudiaban en las distintas oleadas de la pandemia de coronavirus. Desde entonces, tanto el virus como la susceptibilidad de las personas a él han cambiado significativamente.
Los investigadores compararon los genotipos HLA-I de pacientes con COVID-19 en la primera y tercera ola de la pandemia. Se analizaron los genomas de pacientes con COVID-19, incluidos 147 pacientes de la primera ola (entre mayo y agosto de 2020) y 219 pacientes de la tercera ola (entre junio y julio de 2021).
Los investigadores realizaron la tipificación de HLA mediante secuenciación de próxima generación (NGS) que identifica las formas variantes de genes (alelos) en un individuo en particular. Luego, los científicos compararon la frecuencia de aparición de alelos entre los grupos de pacientes.
Encontraron la mitad de pacientes con el alelo HLA-A*01:01 en la tercera ola que en la primera ola. Otras variantes de los genes HLA-I fueron igualmente comunes en ambos grupos.
Anteriormente, se creía que el alelo HLA-A*01:01 estaba asociado con mayores riesgos de infección y un curso grave de COVID-19. Pero ahora los investigadores sugieren que este alelo podría ser más beneficioso de lo que se pensaba anteriormente. El hecho de que ocurra con mucha menos frecuencia entre los pacientes de la tercera ola puede indicar que los portadores de este alelo han desarrollado una sólida inmunidad de células T contra el COVID-19.
HLA-A*01:01 se une principalmente a péptidos que se originan en la región ORF1ab del genoma del SARS-CoV-2. ORF1ab se considera una parte conservativa del genoma, lo que significa que es menos susceptible a las mutaciones que otras regiones. Presumiblemente, los sistemas inmunitarios de los portadores de HLA-A*01:01 han aprendido a detectar la COVID-19 a pesar de las mutaciones.
«Anteriormente, demostramos que los portadores de HLA-A*01:01 tienen un mayor riesgo de COVID-19 grave. Sin embargo, una proporción significativa de pacientes, incluidos los portadores de HLA-A*01:01, tienen una forma leve o incluso asintomática. de COVID-19», explica Maxim Shkurnikov, director del Laboratorio de Investigación sobre Mecanismos Moleculares de la Longevidad de HSE.
«La razón por la que encontramos una disminución en la cantidad de portadores de este alelo entre los pacientes de la tercera ola puede ser que una gran cantidad de estos pacientes ya habían sido infectados cuando llegó la tercera ola y habían formado una fuerte inmunidad de células T, permitiéndoles evitar la hospitalización esta vez».
Además, las denominadas células T de memoria predominan en expacientes de COVID-19 con el alelo HLA-A*01:01; estas células almacenan información sobre una infección durante mucho tiempo después de que se ha eliminado, lo que les permite generar una respuesta inmunitaria rápida al volver a exponerse.
El artículo se publica en la revista PeerJ.
Los hallazgos del estudio sugieren que los sistemas inmunitarios de los portadores del alelo HLA-A*01:01 tienden a ser más efectivos para recordar y reconocer el COVID-19 independientemente de sus mutaciones. Esto confirma la posibilidad de una predisposición genética a la COVID-19 grave. Estos hallazgos también pueden informar el desarrollo de vacunas COVID-19 efectivas dirigidas a la región del genoma ORF1ab.
Más información:
Maxim Shkurnikov et al, Disminución del alelo HLA-A*01:01 en la población de pacientes con COVID-19 asociada con la abundancia de epítopos no estructurales en el repertorio de células T CD8+, PeerJ (2023). DOI: 10.7717/peerj.14707 En medRxiv: www.medrxiv.org/content/10.110 … 022.07.05.22277214v2
Proporcionado por la Escuela Superior de Economía de la Universidad Nacional de Investigación
Citación: Los investigadores revelan la predisposición genética a la inmunidad contra las nuevas variantes de COVID-19 (2023, 17 de enero) recuperado el 17 de enero de 2023 de https://medicalxpress.com/news/2023-01-reveal-genetic-predisposition-immunity-variants.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.