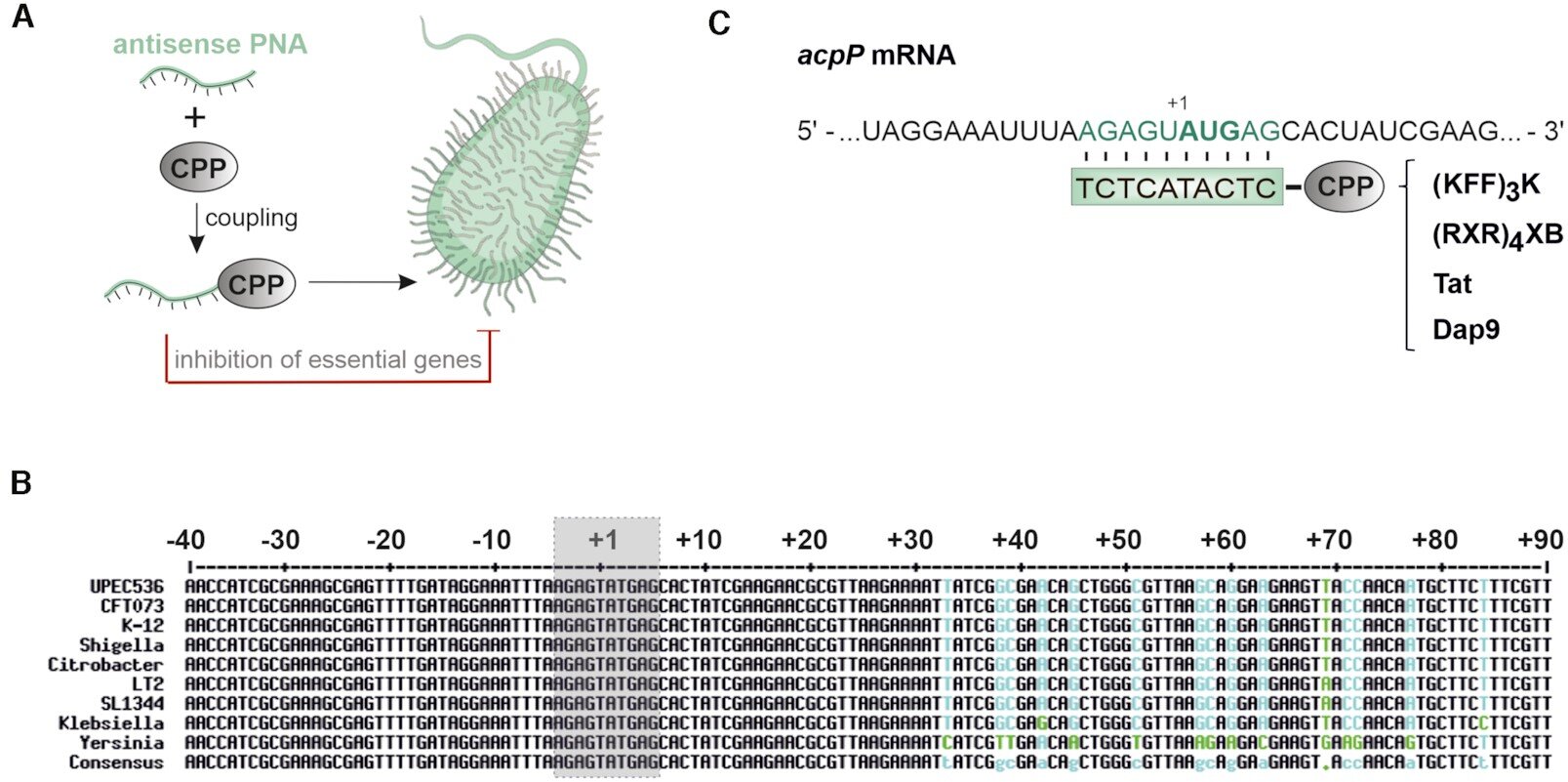
Ilustración esquemática de la tecnología de PNA aplicada y la región diana de PNA del gen esencial conservado, acpP. (A) Un PNA antisentido se acopla a un CPP para facilitar el suministro intrabacteriano. Cuando se dirigen a genes bacterianos esenciales, los conjugados CPP-PNA pueden servir como potentes moléculas antimicrobianas. Partes de esta imagen han sido creadas con Smart (Servier medical art). (B) Alineación de secuencias múltiples (creada con http://multalin.toulouse.inra.fr/multalin/; (118)) del gen esencial acpP que incluye las siguientes proteobacterias γ: UPEC 536 (CP000247.1), E. coli CFT073 (CP051263.1), E. coli K-12 (CP032667.1), Shigella dysenteriae (CP000034.1), Citrobacter rodentium (CP038008.1), Salmonella enterica subsp. enterica serovariedad Typhimurium str. LT2 (AE006468.2), Salmonella enterica subsp. enterica serovariedad Typhimurium str. SL1344 (FQ312003.1), Klebsiella pneumoniae (CP003200.1) y Yersinia pestis (NC_003143.1). Se muestra una sección definida (–40 a +90 nt) que incluye la región alrededor del sitio de unión de PNA (recuadro gris). Se ha aplicado el siguiente código de color: consenso perfecto negro, columnas de alineación variable cian (sustitución de nucleótidos verde). (C) Región relevante del ARNm de acpP en UPEC 536, con la secuencia diana de PNA sombreada en verde. El codón de inicio (AUG) se muestra en negrita. A continuación, se muestra la secuencia de PNA (recuadro verde) con los diferentes CPP conjugados para entrega en UPEC ((KFF)3K, (RXR)4XB, Tat, Dap9). Crédito: Investigación de ácidos nucleicos (2022). DOI: 10.1093/nar/gkac362
En la lucha contra la pandemia de COVID-19, las vacunas basadas en ARNm han demostrado de manera impresionante su potencial. Usando esta tecnología, los científicos pudieron desarrollar y comercializar rápidamente vacunas contra el SARS-CoV-2 que han sido extremadamente efectivas para proteger a millones de personas contra la progresión grave de la enfermedad de COVID-19 o incluso la muerte.
Sin embargo, la medicina basada en ARN puede usarse para combatir más que solo virus. Entre otros, también se puede considerar como candidato a un nuevo tipo de antibióticos que se pueden utilizar para tratar infecciones bacterianas de forma personalizada.
Investigadores de la Universidad de Würzburg (JMU) han investigado qué requisitos previos deben cumplir estos agentes activos y cómo funcionan en la bacteria. Participaron equipos del Instituto de Biología Molecular de Infecciones (IMIB) y del Instituto Helmholtz para la Investigación de Infecciones basada en ARN (HIRI). Presentan los resultados de su trabajo en el número actual de Investigación de ácidos nucleicos.
Los antibióticos tradicionales están fallando cada vez más
«La cantidad de cepas bacterianas resistentes a los antibióticos está creciendo en todo el mundo; los tratamientos con agentes activos convencionales están fallando cada vez más. Por lo tanto, necesitamos urgentemente nuevos medicamentos para combatir estos patógenos de manera dirigida y efectiva», dice el profesor Jörg Vogel. Vogel es el Presidente de Biología Molecular de Infecciones I en JMU y Director de HIRI, así como el autor correspondiente de este estudio.
Los antibióticos de ARNm programables podrían ser la solución a este problema. La estrategia es simple: «Introducimos cadenas cortas de bases en bacterias que están diseñadas para coincidir exactamente con genes específicos», explica Vogel. Cuando estos fragmentos se unen al ARNm correspondiente del gen de interés, anulan la producción de proteínas e, idealmente, la bacteria muere como resultado.
Apagado por la imagen del espejo
En ciencia, este enfoque se conoce como «tecnología antisentido». La estructura de esos agentes activos es una imagen especular de un gen, lo que les permite bloquearlo de manera efectiva. Ya están en el mercado los primeros fármacos que funcionan mediante este principio, como los destinados al tratamiento de las secuelas de la atrofia muscular espinal o la infección por hepatitis C. Sin embargo, los antibióticos de ARNm se han confinado al laboratorio hasta ahora.
En su estudio, los científicos de Würzburg se centraron en cepas bacterianas del tipo Escherichia coli uropatógena (UPEC). En la gran mayoría de los casos, estas bacterias causan una infección del tracto urinario en aproximadamente una de cada dos mujeres una vez en su vida. El uso excesivo de antibióticos durante las últimas décadas ha llevado al desarrollo de resistencias a las terapias actuales en muchas de estas bacterias, complicando particularmente el tratamiento de infecciones recurrentes del tracto urinario que ocurren con frecuencia.
Respuestas a tres preguntas clave
Los equipos de investigación involucrados tenían como objetivo responder a tres preguntas centrales. Primero: ¿Son específicos los agentes activos diseñados (en particular, los ácidos nucleicos peptídicos antisentido que se dirigen a los ARNm de genes bacterianos esenciales)? En otras palabras, ¿realmente bloquean solo un gen bacteriano específico? ¿O es posible que también afecten a otros ARNm? La respuesta es clara: «Nuestros resultados muestran que los pares de bases aplicados solo bloquean el gen de interés», dice Vogel.
Segundo: ¿Cómo reacciona la bacteria a la translocación de estos antibióticos de ARN en la célula? Respuesta: Las bacterias muestran una respuesta al estrés y, por lo tanto, desafortunadamente, no como se deseaba. Esto se debe principalmente al hecho de que los ácidos nucleicos peptídicos antisentido tienen un tamaño comparativamente grande. Por tanto, el estrés se produce principalmente cuando estas biomoléculas atraviesan la membrana bacteriana.
Sin embargo, hay buenas noticias con respecto a la respuesta a la pregunta tres: ¿es posible hacer estos «fragmentos de pares de bases» más pequeños? Sí, lo es. «Hasta ahora, los científicos han asumido que entre nueve y 14 pares de bases son necesarios para evitar cualquier unión no específica a otros genes», explica Vogel. Los resultados publicados ahora muestran que nueve pares de bases son suficientes; por lo tanto, los fragmentos se pueden mantener relativamente pequeños.
En general, según los autores del estudio, los resultados muestran que los antibióticos basados en ARNm son, en principio, adecuados para combatir las cepas uropatógenas de Escherichia coli. Sin embargo, aún deben abordarse varias cuestiones importantes antes de utilizar este enfoque en las clínicas. Aún así, existe una necesidad urgente: «Si no queremos que los microbios resistentes a los antibióticos frustren los éxitos de la medicina moderna, necesitamos herramientas novedosas que faciliten el tratamiento dirigido de los patógenos», dice Jörg Vogel.
Nueva terapia de hipertermia con microondas in situ para superar las infecciones por E. coli multirresistentes
Linda Popella et al, Análisis completo de antibióticos antisentido basados en PNA dirigidos a varios genes esenciales en Escherichia coli uropatógena, Investigación de ácidos nucleicos (2022). DOI: 10.1093/nar/gkac362
Citación: Investigating mRNA-based antibiotics (28 de junio de 2022) recuperado el 29 de junio de 2022 de https://medicalxpress.com/news/2022-06-mrna-based-antibiotics.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.