Los microorganismos presentes en el tracto digestivo del animal podrían tener estrategias moleculares únicas para despolimerizar esta biomasa. Crédito: Gabriela Félix Persinoti
Convertir los residuos agroindustriales en moléculas de interés para la sociedad, como biocombustibles y bioquímicos, es una de las formas de mitigar la dependencia del petróleo y otros combustibles fósiles. Como uno de los mayores productores de biomasa vegetal del mundo, Brasil está bien posicionado para liderar esta transición, pero las materias primas lignocelulósicas (que contienen lignina, hemicelulosa y celulosa) son difíciles de deconstruir o (en términos más técnicos) recalcitrantes a la degradación microbiana y enzimática. .
Los científicos en Brasil están mirando a la naturaleza para descubrir cómo las estrategias para aumentar la disponibilidad de los azúcares que contienen estos materiales pueden mejorar su despolimerización. En Campinas (estado de São Paulo), un grupo de investigación del Laboratorio Nacional de Biorenovables de Brasil (LNBR), brazo del Centro Brasileño de Investigación en Energía y Materiales (CNPEM), realizó un estudio interdisciplinario que involucra ómicas (genómica, proteómica, metabolómica, etc.) y luz de sincrotrón, y descubrió dos nuevas familias de enzimas con potencial biotecnológico producidas por microorganismos en el intestino de capibaras.
Ambas familias de enzimas actúan sobre los componentes de las paredes celulares de las plantas y, por lo tanto, pueden usarse para producir biocombustibles, productos bioquímicos y biomateriales. Uno de ellos también tiene aplicaciones potenciales en la industria láctea porque promueve la degradación de la lactosa.
«Una de nuestras líneas de investigación explora la diversidad brasileña en busca de nuevos mecanismos microbianos que reduzcan la recalcitrancia de los desechos lignocelulósicos. Notamos que el carpincho es un herbívoro altamente adaptado capaz de obtener energía de los desechos vegetales recalcitrantes y que no se ha estudiado mucho mucho», dijo Mário Tyago Murakami, director científico de LNBR y último autor del artículo que informa sobre el estudio publicado en Comunicaciones de la naturaleza.
El carpincho (Hydrochoerus hydrochaeris) es el roedor vivo más grande del mundo y convierte de manera muy eficiente los azúcares contenidos en las plantas en energía, aunque en algunos sectores no le gusta porque puede albergar la garrapata que transmite la fiebre maculosa brasileña, una enfermedad infecciosa rara pero altamente letal. Producida por la bacteria Rickettsia rickettsii.
«Hay muchos estudios sobre rumiantes, especialmente bovinos, pero la información sobre los herbívoros monogástricos es relativamente escasa. A diferencia de los rumiantes, los capibaras digieren el pasto y otras materias vegetales en el ciego, la primera parte del intestino grueso. A la luz de su azúcar altamente eficiente conversión, y porque los capibaras en la región de Piracicaba [of São Paulo state] alimentamos de caña de azúcar, entre otras plantas, partimos de la hipótesis de que los microorganismos presentes en el tracto digestivo del animal podrían tener estrategias moleculares únicas para despolimerizar esa biomasa, que es muy importante para la industria brasileña», dijo Gabriela Felix Persinoti, investigadora en bioinformática del LNBR. y autor correspondiente del artículo.
Metodología novedosa
El enfoque interdisciplinario utilizado en el estudio incluyó multiómica (genómica, transcriptómica y metabolómica utilizadas para caracterizar aspectos moleculares de la microbiota intestinal del carpincho) y bioinformática, así como aceleradores de partículas de CNPEM para analizar las enzimas descubiertas a nivel atómico. «No recuerdo ningún estudio que haya combinado todas estas técnicas, incluido el uso de luz de sincrotrón [a source of extremely bright electromagnetic radiation that helps scientists observe the inner structures of materials]», dijo Murakami. «En esta investigación, nuestro análisis profundizó desde la comunidad microbiana hasta la estructura atómica de ciertas proteínas».
Los científicos analizaron muestras recolectadas del ciego y recto de tres capibaras hembras sacrificadas en Tatuí (estado de São Paulo) en 2017 en el marco de la política local de control de la población de capibaras. Los animales no estaban preñados ni infectados por R. rickettsii.
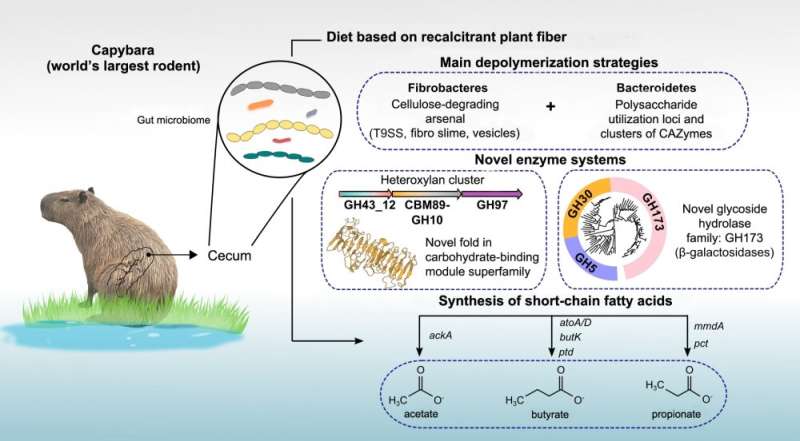
Diagrama esquemático que resume los descubrimientos del estudio. Crédito: Lucelia Cabral et al.
«Las muestras cecales y rectales se recogieron mediante cirugía abdominal. El material se congeló en nitrógeno líquido. Las muestras de ADN y ARN se extrajeron en el laboratorio y se sometieron a secuenciación a gran escala utilizando ómica integradora», dijo Persinoti.
Comenzaron secuenciando genes marcadores, en este caso 16S, presentes en todas las bacterias y arqueas. “Con esta primera secuenciación pudimos detectar diferencias entre las muestras cecales y rectales e identificar en ellas los principales microorganismos. El gen 16S nos dio una respuesta superficial sobre qué microorganismos estaban presentes y abundaban en mayor o menor medida, pero no nos dijo qué enzimas producían los microorganismos o qué enzimas estaban presentes en sus genomas», explicó.
«Para este propósito, utilizamos otra técnica ómica, la metagenómica. Sometimos el ADN de toda la comunidad microbiana en el tracto gastrointestinal de los carpinchos a una secuenciación a gran escala, obteniendo una mayor cantidad de datos. Al implementar una serie de herramientas bioinformáticas, pudimos capaces no sólo de identificar los genomas presentes en cada una de las muestras, y los genes de cada uno de los genomas, sino también de saber qué genes eran nuevos y qué microorganismos nunca habían sido descritos, de esta forma pudimos predecir la funciones de los genes que tenían el potencial para ayudar a despolimerizar la biomasa y convertir el azúcar en energía».
Los investigadores también querían saber qué microorganismos estaban más activos en el momento en que se recolectaron las muestras; en otras palabras, qué genes expresaban realmente los microorganismos. Para ello utilizaron la metatranscriptómica, cuya materia prima es el ARN. «Otra técnica que usamos fue la metabolómica, para confirmar qué metabolitos producían los microorganismos», dijo Persinoti. «Al combinar toda esta información de la ómica, la bioinformática y la expresión génica real y potencial, pudimos descifrar el papel de los microorganismos intestinales para lograr una conversión tan eficiente de las fibras vegetales y descubrir qué genes estaban involucrados en el proceso».
Luego analizaron todos estos datos para identificar genes que podrían desempeñar un papel clave en la reducción de la obstinación de las fibras vegetales, centrándose principalmente en objetivos hasta ahora desconocidos. «La estrategia de selección se centró en genomas novedosos con una gran cantidad de genes involucrados en la despolimerización de la biomasa vegetal», dijo Persinoti. «Vimos cómo estos genes estaban organizados en los genomas de los microorganismos y aprovechamos esta información para averiguar si había genes cercanos con funciones desconocidas que podrían estar involucrados en la descomposición de la fibra vegetal recalcitrante. Esto es importante porque guía la búsqueda de nuevos genes, pero solo cuando pudimos demostrar estos resultados experimentalmente en una etapa posterior pudimos establecer la creación de estas nuevas familias de enzimas».
Habiendo identificado a estos candidatos, pasaron a una demostración bioquímica de sus funciones. «Sintetizamos los genes in vitro y los expresamos usando una bacteria para producir las proteínas correspondientes», dijo Persinoti. «Realizamos varios ensayos enzimáticos y bioquímicos para descubrir las funciones de estas proteínas y dónde actuaban. Determinamos las estructuras atómicas de las proteínas usando luz de sincrotrón y otras técnicas. Con esta información funcional y estructural, pudimos hacer otros experimentos para encontrar determinar qué regiones de las proteínas fueron críticas para su actividad y analizar los mecanismos moleculares subyacentes a sus funciones».
Según Murakami, la doble validación aseguró que las nuevas familias estuvieran realmente involucradas. “Seleccionamos un gen no muy similar a uno que habíamos estudiado previamente en el conjunto de secuencias que teóricamente formaban el universo de una familia recién descubierta. Sintetizamos el gen, lo purificamos, lo caracterizamos bioquímicamente y demostramos que la secuencia tenía el mismo propiedades funcionales como la anterior», explicó. «En otras palabras, caracterizamos a un segundo miembro de la nueva familia para estar absolutamente seguros de que estas proteínas efectivamente constituían una nueva familia».
Nuevas enzimas y cócteles
Persinoti reveló que una de las familias recién descubiertas llamada GH173 tiene aplicaciones potenciales en la industria alimentaria, mientras que otra llamada CBM89 está asociada con el reconocimiento de carbohidratos y podría facilitar la producción de etanol de segunda generación a partir de bagazo y paja de caña de azúcar.
Los investigadores también están desarrollando cócteles de enzimas con hongos hiperproductores de enzimas, y las enzimas recién descubiertas podrían incluirse naturalmente en estas plataformas fúngicas. «El descubrimiento de nuevas familias de enzimas se puede integrar con la transferencia de tecnología para apoyar la innovación», dijo Murakami. «En nuestro grupo, estamos muy interesados en explorar este gran tesoro de la biodiversidad brasileña, particularmente para comprender lo que llamamos materia genómica oscura, partes de estas comunidades microbianas complejas con un potencial desconocido. Nuestro centro cuenta con una excelente infraestructura para este propósito y, junto con nuestras asociaciones con universidades públicas, esto ha permitido que se realicen investigaciones competitivas de este tipo en Brasil. De hecho, el 99% del trabajo, desde el diseño conceptual hasta la ejecución, el análisis y la redacción, se realizó aquí. Dada la inmensa riqueza de la biodiversidad brasileña , era de esperar que tuviéramos las condiciones y capacidades para hacer descubrimientos de alto impacto como estos».
La investigación muestra cómo la genética puede contribuir a los avances en la producción de etanol 2G
Lucelia Cabral et al, El microbioma intestinal del roedor vivo más grande alberga sistemas enzimáticos sin precedentes para degradar los polisacáridos vegetales, Comunicaciones de la naturaleza (2022). DOI: 10.1038/s41467-022-28310-y
Citación: Las enzimas descubiertas en la tripa de carpincho podrían acelerar la utilización de desechos agroindustriales (2 de mayo de 2022) consultado el 2 de mayo de 2022 en https://phys.org/news/2022-05-enzymes-capybara-gut-agroindustrial.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.






