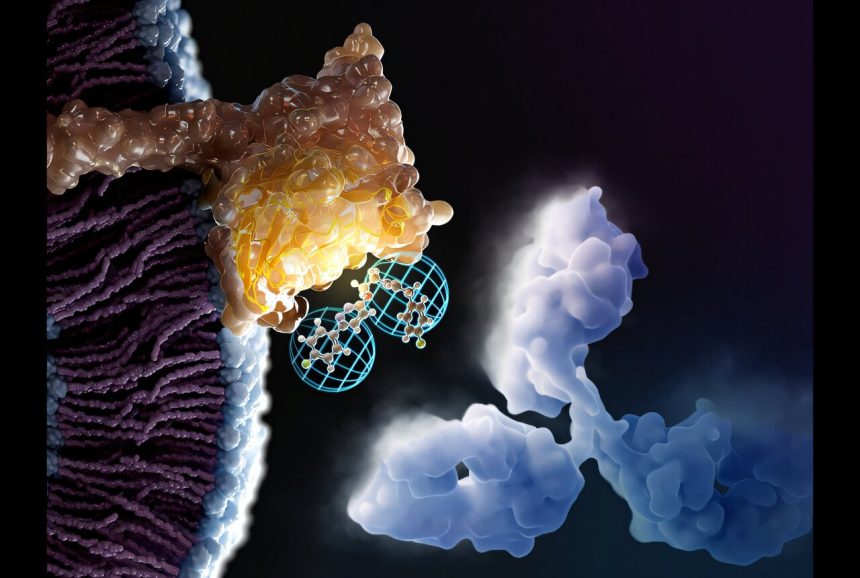
Los investigadores han analizado las interacciones clave entre la proteína del punto de control (amarilla) y un anticuerpo monoclonal (estructura de nube azul claro) para identificar virtualmente pequeñas moléculas que pueden imitar la interacción de los anticuerpos monoclonales con los puntos de control inmunitarios, ofreciendo un enfoque novedoso para la inmunoterapia. Crédito: Laboratorio Gabr
Según los investigadores de Weill Cornell Medicine, el proceso de identificación de candidatos prometedores a fármacos de molécula pequeña dirigidos a los puntos de control del cáncer puede volverse más rápido e inteligente mediante la detección virtual.
Los inhibidores de puntos de control son un tipo de inmunoterapia que trata el cáncer liberando los frenos de las células inmunitarias, de modo que se activan para atacar las células cancerosas. Actualmente, todos los inhibidores de puntos de control aprobados son proteínas grandes fabricadas en laboratorio llamadas anticuerpos monoclonales que deben infundirse en el torrente sanguíneo. Bloquean la unión de las proteínas de punto de control con sus proteínas asociadas, evitando la señal de «apagado» que impide que se activen las células T inmunitarias.
«Nuestra investigación representa un avance notable en el campo de la inmunoterapia contra el cáncer, que puede conducir a una alternativa prometedora a los tratamientos basados en anticuerpos monoclonales», afirmó el autor principal Moustafa Gabr, profesor asistente de química en radiología.
Los hallazgos, publicado 16 de octubre en Avances científicosdemuestran una manera de examinar virtualmente millones de compuestos para encontrar pequeñas moléculas con el potencial de unirse a puntos de control que permitan al sistema inmunológico organizar un ataque contra el cáncer.
Las moléculas pequeñas tienen una ventaja porque generalmente están biodisponibles por vía oral para una administración más fácil, una dosificación personalizada y un mejor cumplimiento del paciente. También pueden penetrar los tumores con mayor eficacia debido a su menor tamaño.
La primera autora del artículo es Somaya Abdel-Rahman, profesora asistente de farmacia en el departamento de química medicinal de la Universidad Mansoura, Egipto.
La dificultad para atacar las proteínas de los puntos de control inmunológico son sus superficies planas y dinámicas, que no se unen fácilmente a las moléculas pequeñas. Para abordar este problema, Gabr y Abdel-Rahman observaron las estructuras cocristalinas de los puntos de control inmunológico unidos a anticuerpos monoclonales.
Los investigadores utilizaron una herramienta computacional que identifica sitios clave de interacción dentro de las interfaces proteína-proteína de las proteínas de punto de control unidas a anticuerpos monoclonales. Esto les ayudó a generar mapas «farmacóforos», modelos tridimensionales que muestran las características esenciales de las interacciones de unión. Luego calcularon propiedades como la fuerza con la que se unen las moléculas y las puntuaciones de «drogabilidad», que son esenciales para el diseño racional de inhibidores de moléculas pequeñas.
Este análisis guió la selección virtual de una base de datos que incluye millones de moléculas pequeñas disponibles comercialmente. Los posibles impactos identificados imitaron las interacciones de unión de los anticuerpos monoclonales con sus puntos de control objetivo.
Los compuestos más prometedores se probaron más a fondo para confirmar la afinidad de unión (qué tan estrechamente interactuaba la molécula con la proteína del punto de control) y la especificidad (la capacidad del compuesto para unirse solo al objetivo previsto). También probaron los compuestos en células para evaluar el impacto funcional de las moléculas pequeñas en la actividad de los puntos de control inmunológico.
Este estudio identificó una molécula llamada MG-T-19, que inhibía fuertemente TIM-3, un receptor de punto de control expresado por una amplia variedad de células inmunes. TIM-3 es un punto de control inhibidor que, cuando se bloquea, puede revitalizar las respuestas de las células T contra los tumores.
Otro compuesto, MG-V-53, fue identificado como un inhibidor de VISTA con actividad antitumoral in vivo. VISTA es otro punto de control inhibidor implicado en la evasión inmune del tumor.
«Las pequeñas moléculas identificadas en este estudio no sólo exhiben fuertes afinidades de unión sino que también muestran una actividad prometedora en modelos de ratón, incluida la reducción del volumen de los tumores», dijo Gabr. Esto sugiere su potencial para el desarrollo terapéutico».
Los estudios futuros se centrarán en optimizar los compuestos de mayor éxito para uso clínico, explorar sus efectos sinérgicos en terapias combinadas y ampliar este enfoque de detección único a objetivos inmunológicos adicionales.
«Este enfoque tiene el potencial de ampliar el repertorio de terapias contra el cáncer disponibles, mejorar los resultados de los pacientes y abrir nuevas vías para la investigación y el desarrollo», afirmó Gabr.
Más información:
Somaya A. Abdel-Rahman et al, Pequeñas moléculas de farmacóforos de anticuerpos (SMAbP) como flujo de trabajo de identificación de hits para puntos de control inmunológico, Avances científicos (2024). DOI: 10.1126/sciadv.adq5540
Citación: El cribado virtual identifica posibles fármacos de molécula pequeña para la inmunoterapia contra el cáncer (2024, 8 de noviembre) recuperado el 10 de noviembre de 2024 de https://medicalxpress.com/news/2024-11-virtual-screening-potential-small-molecule.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.