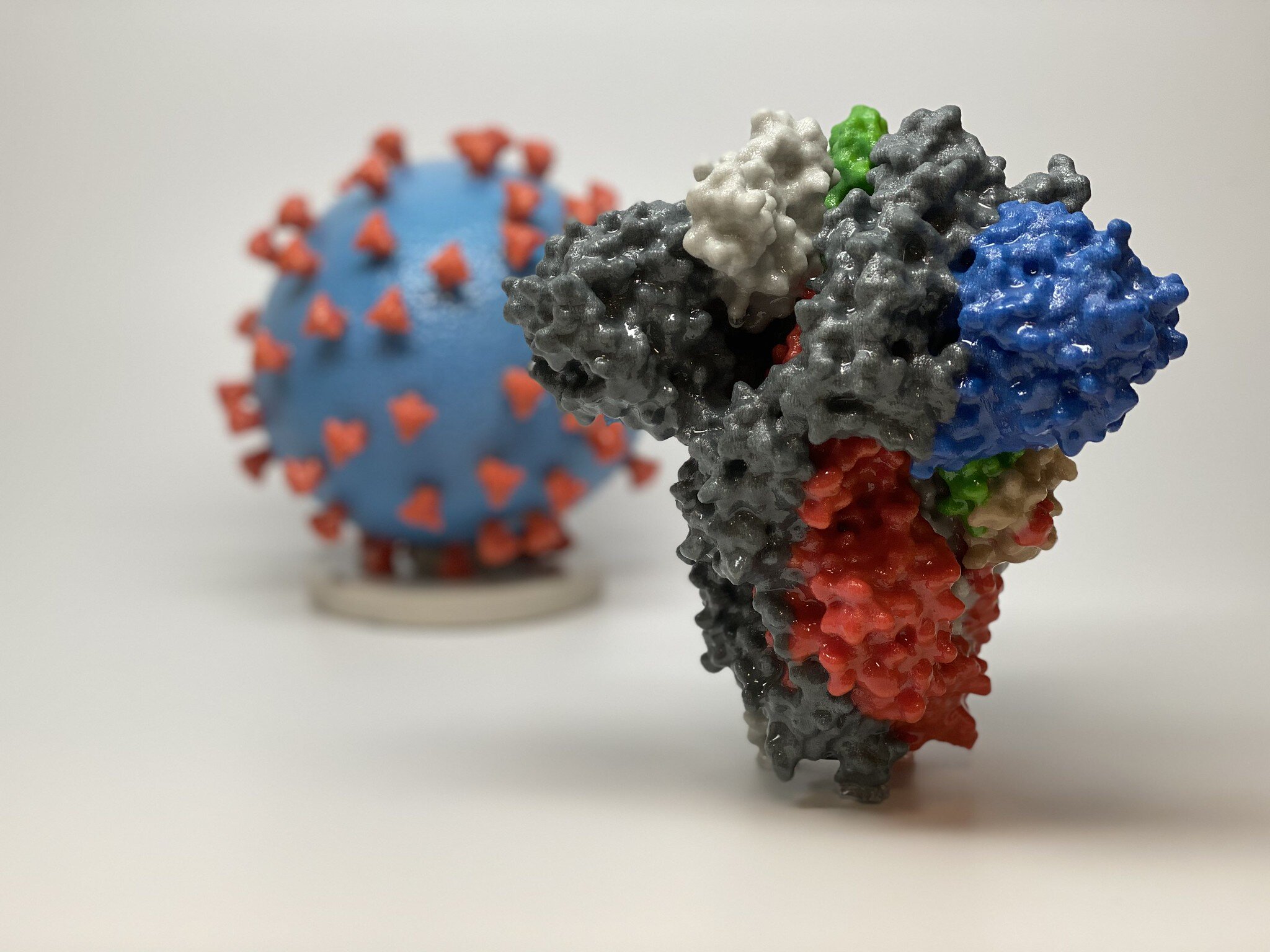
Impresión 3D de una proteína espiga de SARS-CoV-2, el virus que causa el COVID-19, frente a una impresión 3D de una partícula del virus SARS-CoV-2. La proteína espiga (en primer plano) permite que el virus ingrese e infecte las células humanas. En el modelo de virus, la superficie del virus (azul) está cubierta con proteínas de pico (rojo) que permiten que el virus ingrese e infecte las células humanas. Crédito: NIH
Los coronavirus son resbaladizos y eso dificulta la creación de vacunas efectivas que brinden protección a largo plazo. Ahora, los investigadores de la Universidad de Connectiut (UConn) han desarrollado una nueva forma de modelar la proteína de punta del virus y probar su unión a los anticuerpos. Eso podría dar a los científicos un control más firme sobre el virus que causa el COVID-19.
A estas alturas, la mayoría de los ciudadanos adultos en los EE. UU. han sido vacunados contra el COVID y muchos también han recibido vacunas de refuerzo. A pesar de esto, alrededor del 30 % de los pacientes hospitalizados con COVID en Connecticut están completamente vacunados. Aunque hay varias vacunas diferentes contra el COVID en uso en todo el mundo, ninguna de ellas proporciona una protección duradera a largo plazo contra el virus.
El problema es la proteína espiga del coronavirus. Los picos cubren el virus y son tan ondulados y flexibles que se deslizan a través de las garras de los anticuerpos como una comadreja a través de un anillo de bodas. Sin un control firme sobre la espiga, el sistema inmunitario no puede producir buenos anticuerpos. Y sin buenos anticuerpos neutralizantes, la memoria del virus del sistema inmunitario es demasiado borrosa para durar.
«Está un poco fuera de foco. Cualquier cosa fuera de foco es difícil de entender, difícil de entender y difícil de agarrar», dice Paulo Verardi, patobiólogo de la Facultad de Agricultura, Salud y Recursos Naturales de la UConn.
Verardi dio varias conferencias sobre el coronavirus al principio de la pandemia como parte de «La pandemia de COVID-19: impactos en la salud, los negocios y la sociedad», la clase más grande y popular jamás impartida en UConn, y muchos profesores la tomaron. Anna Tarakanova, biofísica computacional y profesora asistente de ingeniería mecánica, fue una de ellas. Es experta en el modelado de proteínas dinámicas. Habló con dos de sus estudiantes de posgrado, Genny Kunkel y Mohammad Madani, y tuvieron una idea. ¿Qué pasaría si modelaran la proteína espiga, descubrieran cómo se movía cuando interactuaba con el sistema inmunitario y luego la congelaban en la posición correcta para que el sistema inmunitario la controlara?
Tarakanova se acercó a Verardi y le gustó la idea. Aunque todas las vacunas COVID actuales aprobadas en los EE. UU. usan versiones estabilizadas de la proteína de pico, nadie sabe si hay posiciones mejores o peores para que el sistema inmunitario las use como plantilla. El equipo de Tarakanova podría averiguarlo. El bioquímico de UConn, Simon White, también se unió al equipo.
Su primer artículo, que describe qué partes de la proteína del pico se flexionan, cómo las mutaciones afectan esa flexibilidad y cómo afecta todo el agarre de los anticuerpos, se publicó en el Revista biofísica el 21 de diciembre. También produjeron videoclips de cómo se mueve la proteína espiga.
«Estamos construyendo activamente modelos de algunas de las variantes naturales emergentes como delta, lambda y omicron para ver cómo evoluciona», dice Tarakanova.
Quieren ver cómo interactúa cada variante con los anticuerpos y, eventualmente, usar ese conocimiento para hacer la mejor versión posible para que los anticuerpos se fijen, de modo que el mundo tenga una vacuna contra COVID que funcione a largo plazo y no requiera un refuerzo cada seis meses.
Genevieve Kunkel et al, Modelado de la dinámica de proteínas de pico de coronavirus: implicaciones para la inmunogenicidad y el escape inmunológico, Revista biofísica (2021). DOI: 10.1016/j.bpj.2021.11.009
Citación: Atrapar el meneo de COVID: los investigadores desarrollan una nueva forma de visualizar cómo la proteína espiga muestra sus movimientos (11 de enero de 2022) recuperado el 11 de enero de 2022 de https://phys.org/news/2022-01-covid-wiggle-visualize -spike-protein.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.