Investigadores del GIST desarrollan un modelo de aprendizaje profundo para predecir interacciones adversas entre fármacos. Utilizando datos de expresión génica, el nuevo modelo puede predecir cómo algunas interacciones farmacológicas pueden provocar efectos adversos en el cuerpo humano. Crédito: Instituto de Ciencia y Tecnología de Gwangju (GIST)
La prescripción de múltiples fármacos, o polifarmacia, suele recomendarse para el tratamiento de enfermedades complejas. Sin embargo, tras la ingestión, varios fármacos pueden interactuar de forma no deseada, lo que puede provocar efectos adversos graves o una disminución de la eficacia clínica. Por lo tanto, la detección temprana de tales interacciones farmacológicas (DDI) es esencial para evitar que los pacientes experimenten efectos adversos.
Actualmente, los modelos computacionales y los algoritmos basados en redes neuronales examinan registros anteriores de interacciones medicamentosas conocidas e identifican las estructuras y los efectos secundarios con los que están asociados. Estos enfoques asumen que medicamentos similares tienen interacciones similares e identifican combinaciones de medicamentos asociadas con efectos adversos similares.
Aunque la comprensión de los mecanismos de los DDI a nivel molecular es esencial para predecir sus efectos indeseables, los modelos actuales se basan en las estructuras y propiedades de los fármacos, con un rango predictivo limitado a las interacciones observadas previamente. No consideran el efecto de los DDI en los genes y la funcionalidad celular.
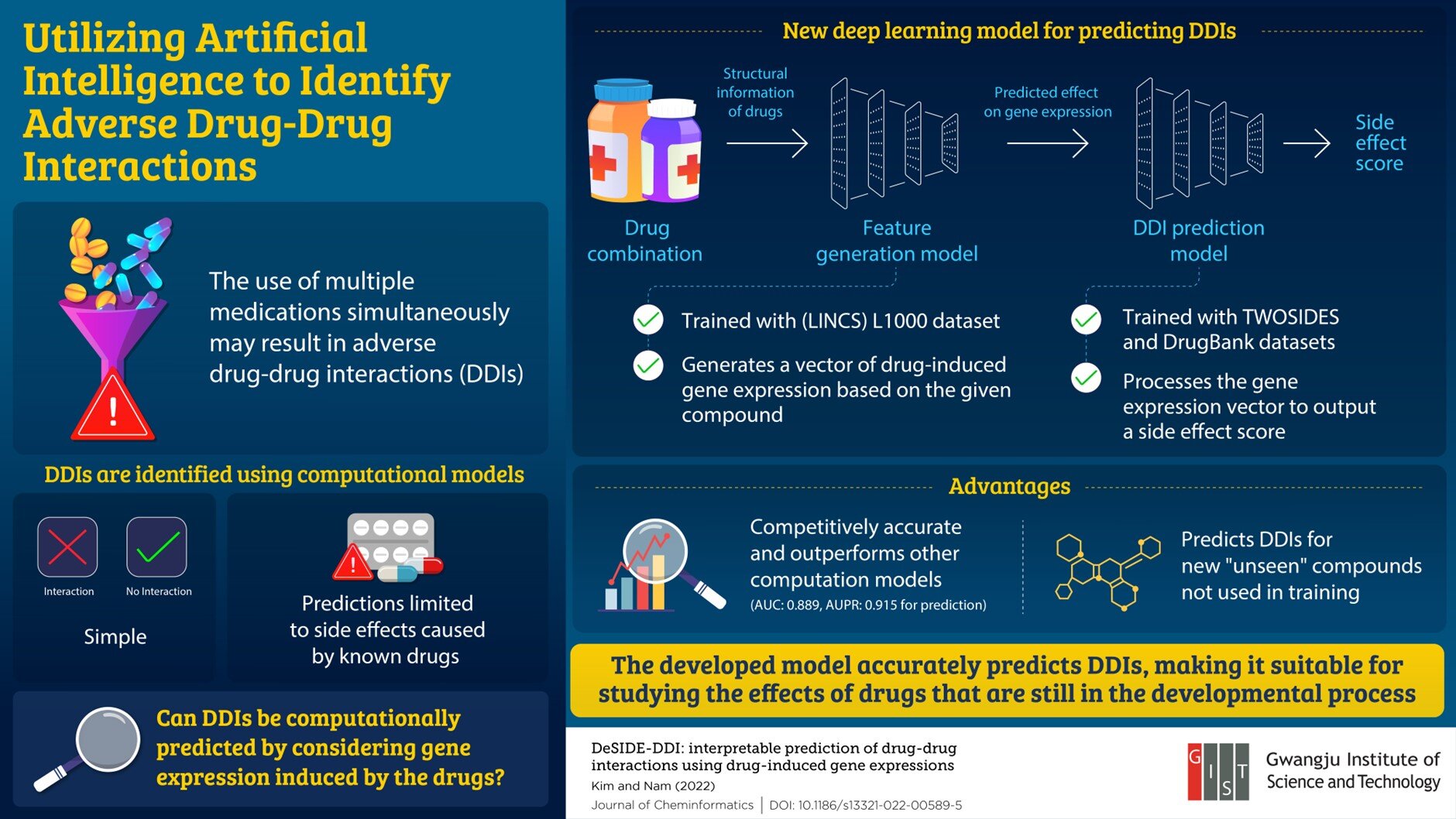
Para abordar estas limitaciones, el profesor asociado Hojung Nam y Ph.D. La candidata Eunyoung Kim del Instituto de Ciencia y Tecnología de Gwangju en Corea del Sur desarrolló un modelo basado en el aprendizaje profundo para predecir las DDI en función de las firmas de expresión génica inducidas por fármacos. Estos hallazgos fueron publicados en el Revista de quimioinformática el 4 de marzo de 2022.
El modelo DeSIDE-DDI consta de dos partes: un modelo de generación de características y un modelo de predicción DDI. El modelo de generación de características predice el efecto de un fármaco sobre la expresión génica considerando tanto la estructura como las propiedades del fármaco, mientras que el modelo de predicción DDI predice varios efectos secundarios resultantes de las combinaciones de fármacos.
Para explicar las características clave de este modelo, el profesor Nam explica: «Nuestro modelo considera los efectos de los medicamentos en los genes mediante el uso de datos de expresión génica, lo que proporciona una explicación de por qué un determinado par de medicamentos causa DDI. Puede predecir DDI para medicamentos aprobados actualmente. medicamentos, así como para nuevos compuestos. De esta manera, las amenazas de la polifarmacia pueden resolverse antes de que los nuevos medicamentos estén disponibles para el público».
Además, dado que todos los compuestos no tienen firmas de expresión génica tratadas con fármacos, este modelo utiliza un modelo de generación de compuestos preentrenado para generar las expresiones génicas tratadas con fármacos esperadas.
Hablando de sus aplicaciones en la vida real, el profesor Nam comenta que «este modelo puede discernir pares de fármacos potencialmente peligrosos, actuando como un sistema de monitoreo de seguridad de fármacos. Puede ayudar a los investigadores a definir el uso correcto del fármaco en la fase de desarrollo del fármaco».
Los verificadores de interacciones de medicamentos podrían haber detectado eventos adversos
Eunyoung Kim et al, DeSIDE-DDI: predicción interpretable de interacciones fármaco-fármaco utilizando expresiones génicas inducidas por fármacos, Revista de quimioinformática (2022). DOI: 10.1186/s13321-022-00589-5
Proporcionado por GIST (Instituto de Ciencia y Tecnología de Gwangju)
Citación: Los investigadores desarrollan un modelo de aprendizaje profundo para predecir interacciones adversas entre fármacos (4 de mayo de 2022) consultado el 4 de mayo de 2022 en https://medicalxpress.com/news/2022-05-deep-adverse-drug-drug-interactions.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.