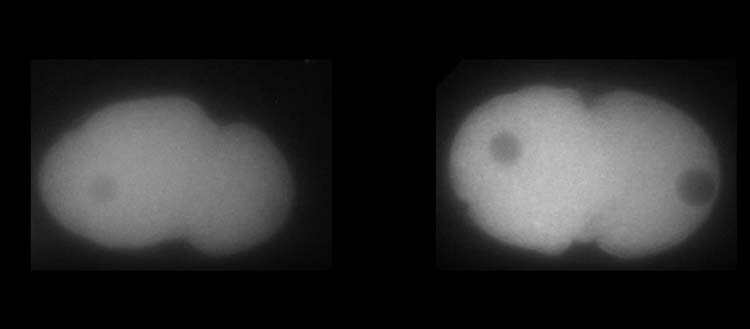
Izquierda, proteína MEX-5. Derecha, proteína PLK-1 en embriones de C. elegans. El blanco muestra la mayor o menor presencia de las proteínas. Crédito: UNIGE / Laboratoire Monica Gotta
Cada una de nuestras células contiene alrededor de 40 millones de proteínas que juntas realizan todas las tareas que la célula necesita para sobrevivir. Para una acción suave, las proteínas adecuadas deben concentrarse en cantidades específicas, en un momento específico y en un lugar específico. Sin embargo, establecer una distribución tan delicada requiere un proceso extremadamente preciso, que ocurre a resoluciones espaciales minúsculas que las herramientas estándar de biología celular a menudo no pueden detectar. Para comprender cómo funciona este mecanismo, investigadores de la Universidad de Ginebra (UNIGE) desarrollaron un nuevo enfoque que combina experimentos de genética y biología celular con modelos físicos. Utilizando algoritmos específicos, simularon la formación de gradientes de proteínas en 3D ya lo largo del tiempo y pudieron explicar estos complejos mecanismos. Además, su modelo innovador se puede adaptar a otros sistemas biológicos para investigar la dinámica de las proteínas. Estos resultados se pueden leer en el procedimientos de la Academia Nacional de Ciencias.
Como una gota de tinta en un vaso de agua, las proteínas pueden difundirse y distribuirse uniformemente por toda la célula. Sin embargo, para bastantes tareas, las proteínas necesitan formar gradientes. «Los gradientes de proteínas, que surgen de la distribución desigual de proteínas en áreas celulares específicas, son fundamentales para muchas funciones celulares y del organismo», explica Monica Gotta, profesora del Departamento de Fisiología Celular y Metabolismo y del Centro de Investigación Traslacional en Onco- hematología (CRTOH) de la Facultad de Medicina de la UNIGE, quien dirigió este trabajo. «Por ejemplo, los gradientes de proteínas son importantes para la diferenciación celular, el proceso por el cual los diferentes tipos de células que constituyen un organismo complejo emergen de una única célula, el óvulo fertilizado».
Un uso de la aleatoriedad
Se sabe que la proteína PLK-1, un regulador clave de la división celular, está más concentrada en el lado anterior del embrión. Pero, ¿cómo se puede implementar este mecanismo y cuál sería la consecuencia si el más mínimo detalle fallara? Como las herramientas habituales de la biología no eran suficientes para responder a esta pregunta, Monica Gotta se alegró de dar la bienvenida a su equipo a la física, Sofia Barbieri, investigadora postdoctoral en el Departamento de Fisiología Celular y Metabolismo de la Facultad de Medicina de UNIGE.
“Compilando todo lo conocido sobre este proceso biológico y nuevas hipótesis sobre los mecanismos, desarrollé un modelo estadístico de formación de gradientes de proteínas basado en matemáticas probabilísticas”, explica Sofia Barbieri. «Recurrí a algoritmos computacionales específicos, llamados simulaciones de Monte Carlo, que llevan el nombre de la famosa ciudad de juego». Estos algoritmos se utilizan para modelar fenómenos con un alto nivel de complejidad, como las finanzas, el comercio o la física de partículas.
El equipo pudo simular gradientes de proteínas, no solo en 3D, sino también a lo largo del tiempo. Sin embargo, dicho modelo requería varias iteraciones entre la optimización de parámetros y la comparación con datos biológicos. Los investigadores construyeron una primera versión del modelo incorporando todos los elementos físicos y biológicos conocidos del sistema, luego introdujeron los parámetros específicos necesarios para probar varias hipótesis sobre las variables desconocidas. Simularon posibles resultados físicos y biológicos que reproducían computacionalmente la dinámica de proteínas y el establecimiento de gradientes en la célula, y los probaron en la vida real con experimentos in vivo utilizando los embriones de un pequeño gusano, el nematodo C. elegans.
Intrincadas interacciones de proteínas en juego
Gracias a la interacción continua entre el modelado y la biología celular, los investigadores de UNIGE pudieron determinar cómo se establecía y mantenía el gradiente de PLK-1 a lo largo del tiempo. De hecho, PLK-1 debe unirse y desvincularse dinámicamente de MEX-5, otra proteína crucial para el desarrollo en el embrión de C. elegans, para contrarrestar su tendencia natural a difundirse homogéneamente en la célula. De hecho, MEX-5 tiene la capacidad de cambiar su difusividad dependiendo de su posición dentro de la célula y de interactuar con otras proteínas, lo cual es esencial para enriquecer PLK-1 donde sea necesario. «Sin embargo, sorprendentemente, MEX-5 no es tan eficiente en su tarea, ya que una gran cantidad de PLK-1 no está ligada a MEX-5», señala Sofia Barbieri.
Este estudio proporciona un modelo cuantitativo único para comprender las interacciones dinámicas entre proteínas y se puede adaptar a otras células o proteínas cuyos mecanismos complejos no se pueden probar con los experimentos habituales de biología celular. “Nuestro trabajo demuestra que las colaboraciones interdisciplinarias son cada vez más importantes para avanzar en la investigación”, concluye Monica Gotta.
Recuerdos heredados de un sitio cromosómico
El modelado de la dinámica de proteínas en embriones revela que el gradiente de PLK-1 se basa en mecanismos de reacción-difusión débilmente acoplados, procedimientos de la Academia Nacional de Ciencias (2022). DOI: 10.1073/pnas.2114205119.
Citación: Cómo la física y la biología trabajan juntas para explorar los mecanismos de la vida (2022, 7 de marzo) recuperado el 7 de marzo de 2022 de https://phys.org/news/2022-03-physics-biology-explore-mechanisms-life.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.