El ADN antiguo conservado en el sarro dental de fósiles humanos codifica metabolitos microbianos que podrían ser el próximo antibiótico. Crédito: Fundación Werner/Siemens
Los microorganismos, en particular las bacterias, son químicos hábiles que pueden producir una diversidad impresionante de compuestos químicos conocidos como productos naturales. Estos metabolitos brindan a los microbios importantes ventajas evolutivas, como permitirles interactuar entre sí o con su entorno y ayudarlos a defenderse de diferentes amenazas. Debido a las diversas funciones que tienen los productos naturales bacterianos, muchos han sido utilizado como tratamientos médicos como antibióticos y medicamentos contra el cáncer.
Las especies microbianas vivas hoy representan solo una pequeña fracción de la gran diversidad de microbios que han habitado la Tierra durante los últimos 3 mil millones de años. Explorar este pasado microbiano presenta oportunidades emocionantes para recuperar parte de su química perdida.
El estudio directo de estos metabolitos en muestras arqueológicas es virtualmente imposible debido a su mala conservación con el tiempo. Sin embargo, reconstruirlos utilizando los planos genéticos de microbios muertos hace mucho tiempo podría proporcionar un camino a seguir.
Somos un equipo de antropólogos, arqueogenéticos y bioquimicos que estudian microbios antiguos. Por generando compuestos químicos previamente desconocidos a partir de los genomas reconstruidos de bacterias antiguas, nuestra investigación recientemente publicada proporciona una prueba de concepto para el uso potencial de microbios fósiles como fuente de nuevos medicamentos.
Reconstruyendo genomas antiguos
La maquinaria celular que produce productos naturales bacterianos está codificada en genes que normalmente están muy cerca unos de otros, formando lo que se denomina grupos de genes biosintéticos. Dichos genes son difíciles de detectar y reconstruir a partir de ADN antiguo porque el material genético muy antiguo se descompone con el tiempo, fragmentándose en miles o incluso millones de piezas. El resultado final son numerosos fragmentos diminutos de ADN. menos de 50 nucleótidos de largo todo mezclado como un rompecabezas revuelto.

Un solo diente antiguo conserva los genomas de millones de bacterias antiguas. Crédito: Felix Wey/Fundación Werner Siemens
Secuenciamos miles de millones de fragmentos de ADN tan antiguos y luego mejoramos un proceso bioinformático llamado asamblea de novo para ordenar digitalmente los fragmentos de ADN antiguo en tramos de hasta 100.000 nucleótidos de largo, una mejora de 2.000 veces. Este proceso nos permitió identificar no solo qué genes estaban presentes, sino también su orden en el genoma y las formas en que se diferencian de los genes bacterianos conocidos hoy en día, información clave para descubrir su historia evolutiva y función.
Este método nos permitió echar un vistazo sin precedentes a los genomas de microbios que vivieron hace 100.000 años, incluidas especies que no se sabe que existen en la actualidad. Nuestros hallazgos hacen retroceder la previamente más antiguo genomas microbianos reconstruidos por más de 90.000 años.
En los genomas microbianos que reconstruimos a partir del ADN extraído del antiguo sarro dental, encontramos un grupo de genes que compartían una alta proporción de neandertales y humanos anatómicamente modernos que vivieron durante el Paleolítico medio y superior que duró desde hace 300.000 a 12.000 años. Este grupo llevó el características moleculares de ADN muy antiguo y pertenecía al género bacteriano clorobioun grupo de bacterias verdes del azufre capaces de realizar la fotosíntesis.
Insertamos una versión sintética de este grupo de genes en una bacteria «moderna» llamada Pseudomona protegens por lo que podría producir los compuestos químicos codificados en los genes antiguos. Usando este método, pudimos aislar dos compuestos previamente desconocidos que llamamos paleofurano A y B y determinar su estructura química. La resintetización de estas moléculas en el laboratorio desde cero confirmó su estructura y nos permitió producir cantidades más grandes para su posterior análisis.
Al reconstruir estos compuestos antiguos, nuestros hallazgos resaltan cómo las muestras arqueológicas podrían servir como nuevas fuentes de productos naturales.
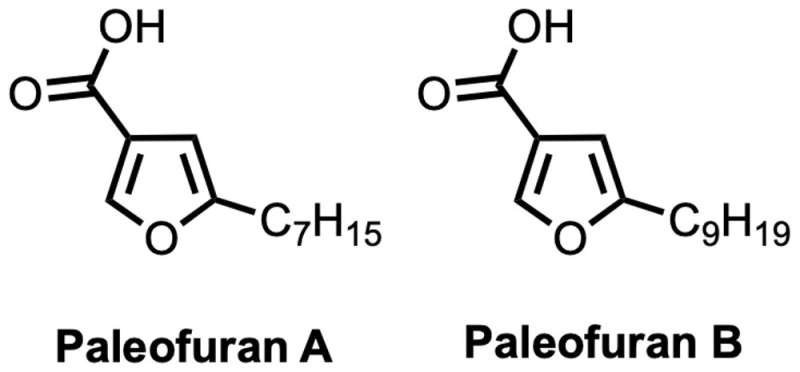
Estos paleofuranos se produjeron a partir de ADN microbiano antiguo. Crédito: Pierre Stallforth, CC POR
Minería de productos naturales antiguos
Los microbios evolucionan constantemente y se adaptan al entorno que los rodea. Debido a que los entornos que habitan hoy en día difieren de los de sus antepasados, es probable que los microbios de hoy produzcan productos naturales diferentes a los de los microbios antiguos de hace decenas de miles de años.
Tan recientemente como Hace 25.000 a 10.000 añosla Tierra experimentó un gran cambio climático a medida que pasaba de un clima más frío y volátil Época del Pleistoceno a las más cálidas y templadas Época del Holoceno. Los estilos de vida humanos también cambiaron drásticamente durante esta transición a medida que las personas comenzaron a vivir fuera de las cuevas y experimentaron cada vez más con la producción de alimentos. Estos cambios los pusieron en contacto con diferentes microbios a través de la agricultura, la cría de animales y sus nuevos entornos construidos. El estudio de las bacterias de la era del Pleistoceno puede arrojar información sobre las especies bacterianas y los genes biosintéticos que ya no están asociados con los humanos en la actualidad, y tal vez incluso los microbios que se han extinguido.
Si bien la cantidad de datos recopilados por los científicos sobre organismos biológicos ha aumentado exponencialmente en las últimas décadas, la cantidad de nuevos antibióticos se ha estancado. Esto es particularmente problemático cuando las bacterias pueden evadir los tratamientos con antibióticos existentes más rápido de lo que los investigadores pueden desarrollar otros nuevos.
Mediante la reconstrucción de genomas microbianos a partir de muestras arqueológicas, los científicos pueden aprovechar la diversidad oculta de productos naturales que de otro modo se habrían perdido con el tiempo, aumentando la cantidad de fuentes potenciales a partir de las cuales pueden descubrir nuevos medicamentos.
Ampliación de moléculas antiguas
Nuestro estudio ha demostrado que es posible acceder a productos naturales del pasado. Para aprovechar la gran diversidad de compuestos químicos codificados en el ADN antiguo, ahora debemos optimizar nuestra metodología para que requiera menos mano de obra.
Actualmente estamos optimizando y automatizando nuestro proceso para identificar genes biosintéticos en ADN antiguo de manera más rápida y confiable. También estamos implementando sistemas robóticos de manejo de líquidos para completar los pasos de pipeteo y cultivo bacteriano que requieren mucho tiempo en nuestros métodos. Nuestro objetivo es ampliar el proceso para poder traducir una gran cantidad de datos sobre microbios antiguos en el descubrimiento de nuevos agentes terapéuticos.
Aunque podemos recrear moléculas antiguas, sus roles biológicos y ecológicos son difíciles de descifrar. Dado que las bacterias que originariamente producían estos compuestos ya no existen, no podemos cultivarlas ni manipularlas genéticamente. El estudio adicional deberá basarse en bacterias similares que se pueden encontrar hoy. Queda por probar si las funciones de estos compuestos han seguido siendo las mismas en los parientes modernos de los microbios antiguos. Aunque las funciones originales de estos compuestos para los microbios antiguos pueden ser desconocidas, todavía tienen el potencial de ser reutilizados para tratar enfermedades modernas.
En última instancia, nuestro objetivo es arrojar nueva luz sobre la evolución microbiana y luchar contra la actual crisis de antibióticos proporcionando un nuevo eje temporal para el descubrimiento de antibióticos.
Este artículo se vuelve a publicar de La conversación bajo una licencia Creative Commons. Leer el artículo original.![]()
Citación: La reconstrucción de genomas bacterianos antiguos puede revivir moléculas previamente desconocidas, una fuente potencial de nuevos antibióticos (6 de mayo de 2023) consultado el 6 de mayo de 2023 en https://phys.org/news/2023-05-reconstructing-ancient-bacterial-genomes- revivir.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.