por Ronja Münch, Instituto Leibniz para la Investigación de Productos Naturales y Biología de Infecciones – Instituto Hans Knoell –
JIPipe se puede utilizar, por ejemplo, para analizar los perfiles de movimiento de los nematodos. Crédito: Hannah Büttner, Zoltán Cseresnyés, Ruman Gerst / Leibniz-HKI
El software JIPipe fue desarrollado por científicos del Instituto Leibniz para la Investigación de Productos Naturales y Biología de Infecciones (Leibniz-HKI) y simplifica significativamente el análisis de las imágenes generadas en la investigación. Los usuarios pueden crear diagramas de flujo de acuerdo con las necesidades de su aplicación y, por lo tanto, realizar análisis automáticos de imágenes utilizando inteligencia artificial sin ningún conocimiento de programación. JIPipe se basa en ImageJ, un programa estándar para el análisis científico de imágenes microscópicas biomédicas. Los autores presentan ahora su desarrollo en Métodos de la naturaleza.
Las imágenes, especialmente las imágenes microscópicas, juegan un papel importante en la investigación biomédica. Con la ayuda de etiquetas fluorescentes, por ejemplo, los procesos en las células se vuelven visibles. «Una imagen vale más que mil palabras, eso sigue siendo cierto», dice Thilo Figge, jefe del grupo de investigación de Biología de Sistemas Aplicados en Leibniz-HKI y profesor de la Universidad Friedrich Schiller de Jena.
Pero el análisis plantea desafíos cada vez mayores para los investigadores. «Se están generando resoluciones cada vez más altas y, por lo tanto, mayores cantidades de datos», explica Figge. «Al mismo tiempo, los métodos de IA, o inteligencia artificial, ahora son tan avanzados que son cada vez más difíciles de usar para investigadores sin conocimientos de programación».
El programa de código abierto JIPipe, abreviatura de Java Image Processing Pipeline, desarrollado en Leibniz-HKI tiene como objetivo simplificar eso. «JIPipe es una herramienta que no requiere ninguna habilidad de programación», explica el desarrollador Ruman Gerst, miembro del grupo de investigación de Biología de Sistemas Aplicados. En cambio, el software utiliza un lenguaje de programación visual: con la ayuda de bloques de construcción prefabricados, los usuarios pueden crear flujos de trabajo individuales para analizar imágenes automáticamente de acuerdo con sus requisitos específicos.
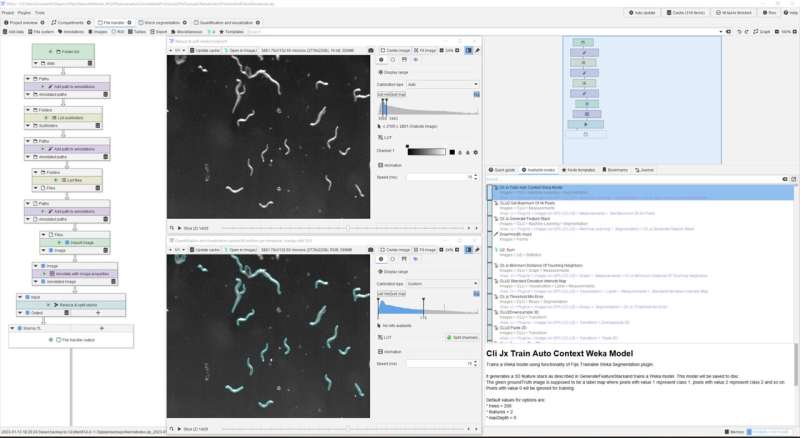
Captura de pantalla de un análisis JIPipe de supervivencia de nematodos. Crédito: Zoltán Cseresnyés/Leibniz-HKI
JIPipe admite otros lenguajes de programación
El programa se basa en el software de código abierto ImageJ, que se ha establecido como un estándar en el análisis científico de imágenes. JIPipe e ImageJ son totalmente compatibles y se complementan entre sí en el análisis científico de imágenes. «Nuestro programa es compatible con los scripts de ImageJ e incluye las funciones y macros habituales», explica Gerst. También se admiten otros lenguajes de programación como Python y R.
El programa precursor fue desarrollado hace varios años por Zoltán Cseresnyés, también miembro del grupo de investigación de Biología de Sistemas Aplicados. «Originalmente, escribí el código para un ensayo de fagocitosis», explica Cseresnyés. En la fagocitosis, una célula toma una partícula, como otra célula, y la descompone, lo que generalmente se visualiza con tintes fluorescentes.
Con el tiempo, el especialista en imágenes siguió ampliando el código para nuevas aplicaciones y el programa se volvió difícil de manejar y demasiado complejo. «Nos dimos cuenta de que teníamos que rediseñarlo y hacerlo modular», dice Cseresnyés, razón por la cual el equipo incorporó al bioinformático Ruman Gerst. También sugirió el lenguaje de programación visual actual, que permite al equipo analizar datos de imágenes de cualquier problema biomédico.
Resultados reproducibles
JIPipe ya se ha utilizado para varios estudios, por ejemplo, para investigar la eficacia de la administración de fármacos mediante los llamados nanoportadores en el hígado o para probar la tasa de supervivencia de los nematodos que han digerido bacterias productoras de toxinas. Con el nuevo programa también se han analizado los enfrentamientos entre las células inmunitarias y las esporas de hongos. Los desarrolladores también ofrecen cursos para su uso como parte del Microverse Imaging Center y NFDI4BioImage, parte de la Infraestructura Nacional de Datos de Investigación de Alemania.
«A diferencia del análisis de imágenes manual, el análisis automatizado siempre proporciona los mismos resultados, por lo tanto, es reproducible y cumple con los llamados principios FAIR para el análisis de imágenes», enfatiza Thilo Figge. El término FAIR se origina en el campo de la gestión de datos de investigación y significa «Findable, Aaccesible, yointeroperable y Rreutilizable».
Más información:
Ruman Gerst et al, JIPipe: procesamiento por lotes visual para ImageJ, Métodos de la naturaleza (2023). DOI: 10.1038/s41592-022-01744-4
Proporcionado por el Instituto Leibniz para la Investigación de Productos Naturales y la Biología de las Infecciones – Instituto Hans Knoell –
Citación: El nuevo software permite el análisis automatizado de datos de imágenes biomédicas sin conocimientos de programación (31 de enero de 2023) consultado el 31 de enero de 2023 en https://phys.org/news/2023-01-software-enables-automated-analysis-biomedical.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.






