Gráficamente abstracto. Crédito: iCiencia (2022). DOI: 10.1016/j.isci.2022.104318
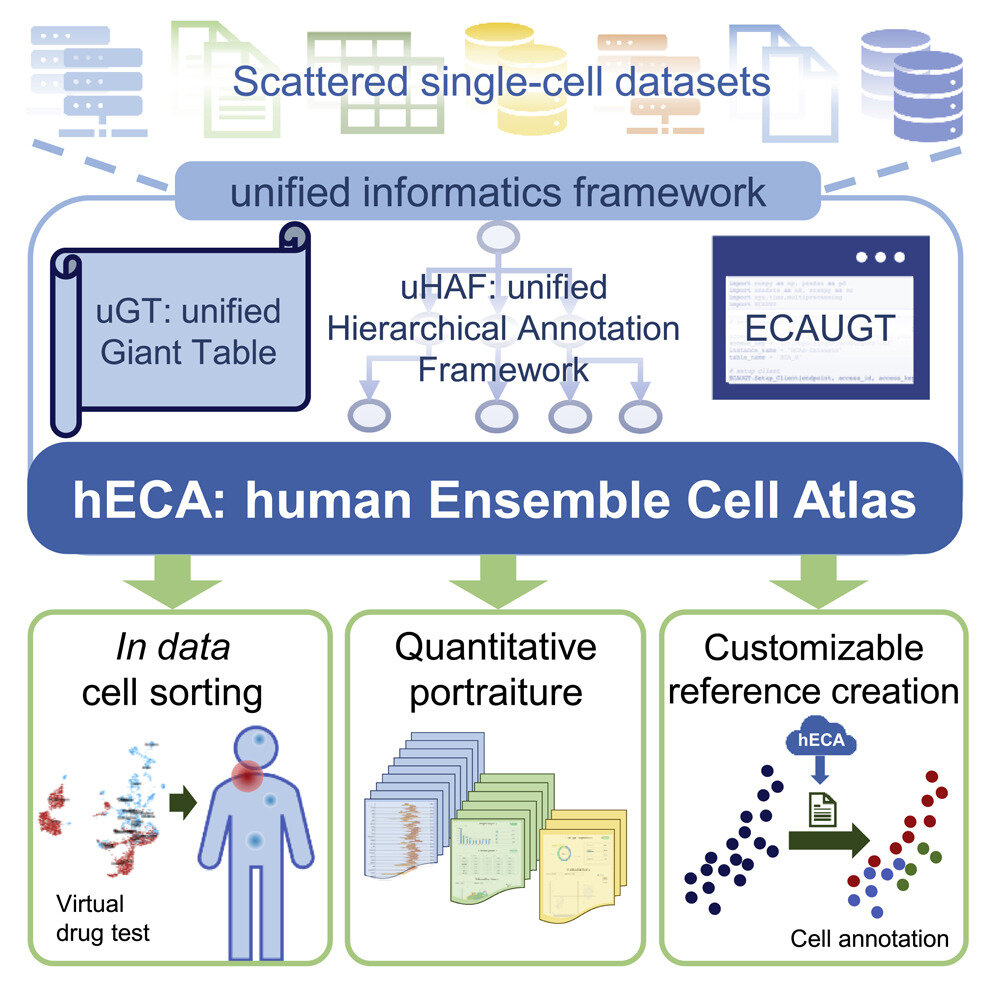
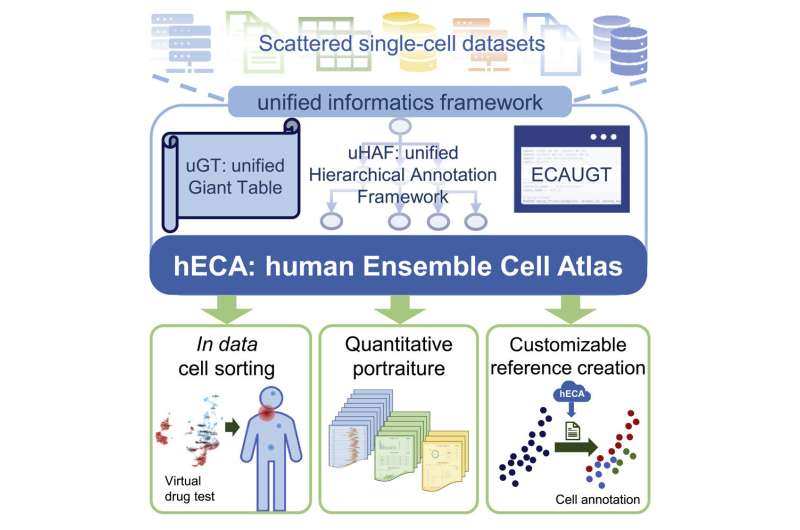
Imagine un cuerpo humano virtual, rico en complejidad y detalle, que permita a los científicos simular experimentos que no se pueden realizar in vivo o in vitro. Un equipo de investigadores chinos acercó esta visión a la realidad mediante el desarrollo de un marco para el ensamblaje continuo de datos centrados en células y construyó el Ensemble Cell Atlas humano (hECA) utilizando datos recopilados de conjuntos de datos públicos dispersos.
Presentaron su marco informático unificado en un estudio publicado el 28 de abril en iCiencia. hECA también ha realizado una contribución histórica a la integración de datos de células individuales humanas de múltiples fuentes y la realización de análisis posteriores, que se publicó en Biología Cuantitativa el 4 de julio.
«Los estudios de caso del hECA demostraron la revolución que un atlas de células de conjunto centrado en las células puede aportar a la investigación biomédica», dijo el autor del estudio, Xuegong Zhang, de la Universidad de Tsinghua.
El rápido desarrollo de las tecnologías de secuenciación de células individuales, especialmente un método de secuenciación de ARN conocido como transcriptómica de células individuales, ha permitido a los científicos perfilar células individuales y examinar qué genes están activados en diferentes tipos de células.
Científicos de todo el mundo están comprometidos en la construcción de «atlas» de resolución de una sola célula de todos los diferentes tipos de células en proyectos como el Atlas de Células Humanas (HCA) y el Programa Atlas BioMolecular Humano. Pero todavía hay cierta incertidumbre sobre cómo debe definirse y ensamblarse un atlas celular.
«El punto clave del ensamblaje del atlas celular es la organización de la información celular», dijo Zhang.
Desde el lanzamiento del proyecto HCA en 2017, se han publicado muchos artículos sobre atlas de células, y la mayoría de ellos son colecciones de una gran variedad de datos de una sola célula documentados e indexados proyecto por proyecto. Estudios anteriores argumentaron que el mapeo celular se trata de crear un esqueleto tridimensional del cuerpo humano y simplemente ensamblar las células observadas en sus posiciones correspondientes. Sin embargo, un cuerpo humano es demasiado complejo para este tipo de montaje.
En cambio, «el ensamblaje de un atlas de celdas debería transmitir la naturaleza multifacética de los datos y permitir a los usuarios buscar con condiciones personalizadas entre diferentes métodos de indexación», dijo Zhang.
Mientras tanto, cantidades masivas de datos transcriptómicos unicelulares están llegando al dominio público a partir de colaboraciones multiinstitucionales, generando petabytes de datos que cubren todos los principales órganos humanos adultos, así como etapas clave de desarrollo o patológicas.
Para el equipo de Zhang, estos datos públicos dispersos de una sola célula sugirieron un enfoque alternativo para construir un atlas de células: comenzar de abajo hacia arriba reuniendo datos de múltiples fuentes.
Para ensamblar datos de esta escala de múltiples fuentes en un atlas conjunto, los investigadores desarrollaron un marco informático unificado, que incluía una infraestructura de base de datos especial para almacenar datos de una sola celda con dimensionalidad y volumen ultra altos, así como un marco de anotación jerárquico unificado. para hacer que las etiquetas de tipo de celda de diferentes conjuntos de datos sean comparables y consistentes. Los investigadores también diseñaron una interfaz de programación de aplicaciones para recuperar eficientemente las células del atlas.
Con estas tecnologías, el equipo desarrolló tres nuevos esquemas para aplicar el atlas ensamblado. Primero, habilitaron la clasificación de celdas de datos para seleccionar celdas del cuerpo humano virtual de celdas ensambladas usando combinaciones flexibles de expresiones lógicas. Crearon un sistema de «retrato cuantitativo» para representar la información completa de genes, tipos de células y órganos. También construyeron una creación de referencia personalizable para que los usuarios personalicen sus referencias para tareas de anotación de tipo de celda.
Los investigadores realizaron una serie de experimentos para verificar e ilustrar la calidad y la usabilidad de los datos ensamblados en múltiples escenarios de aplicación. Los ejemplos de casos incluyeron la investigación de fármacos fuera de los objetivos (consecuencias biológicas no deseadas de un fármaco) en todo el cuerpo, lo que demostró el poder del atlas de células de conjunto para abrir nuevas posibilidades en la investigación biomédica.
Según el estudio, este tipo de clasificación de celdas de datos puede revelar importantes patrones específicos de órganos y ayudar a los científicos a determinar los órganos que son más susceptibles a los efectos secundarios de la terapia con medicamentos dirigidos.
Los investigadores han desarrollado estrategias y tecnologías para integrar más datos de alta calidad de otros conjuntos de datos integrales y continuarán mejorando y actualizando futuras versiones de hECA.
Más información:
Chenwei Li et al, Integración de datos de células individuales humanas de múltiples fuentes, Biología Cuantitativa (2022). DOI: 10.15302/J-QB-022-0304. journal.hep.com.cn/qb/EN/10.15302/J-QB-022-0304
Sijie Chen et al, hECA: El ensamblaje centrado en las células de un atlas celular, iCiencia (2022). DOI: 10.1016/j.isci.2022.104318
Proporcionado por Higher Education Press
Citación: Los investigadores construyen un atlas de células utilizando conjuntos de datos de células individuales dispersos (27 de diciembre de 2022) recuperado el 27 de diciembre de 2022 de https://phys.org/news/2022-12-cell-atlas-single-cell-datasets.html
Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.