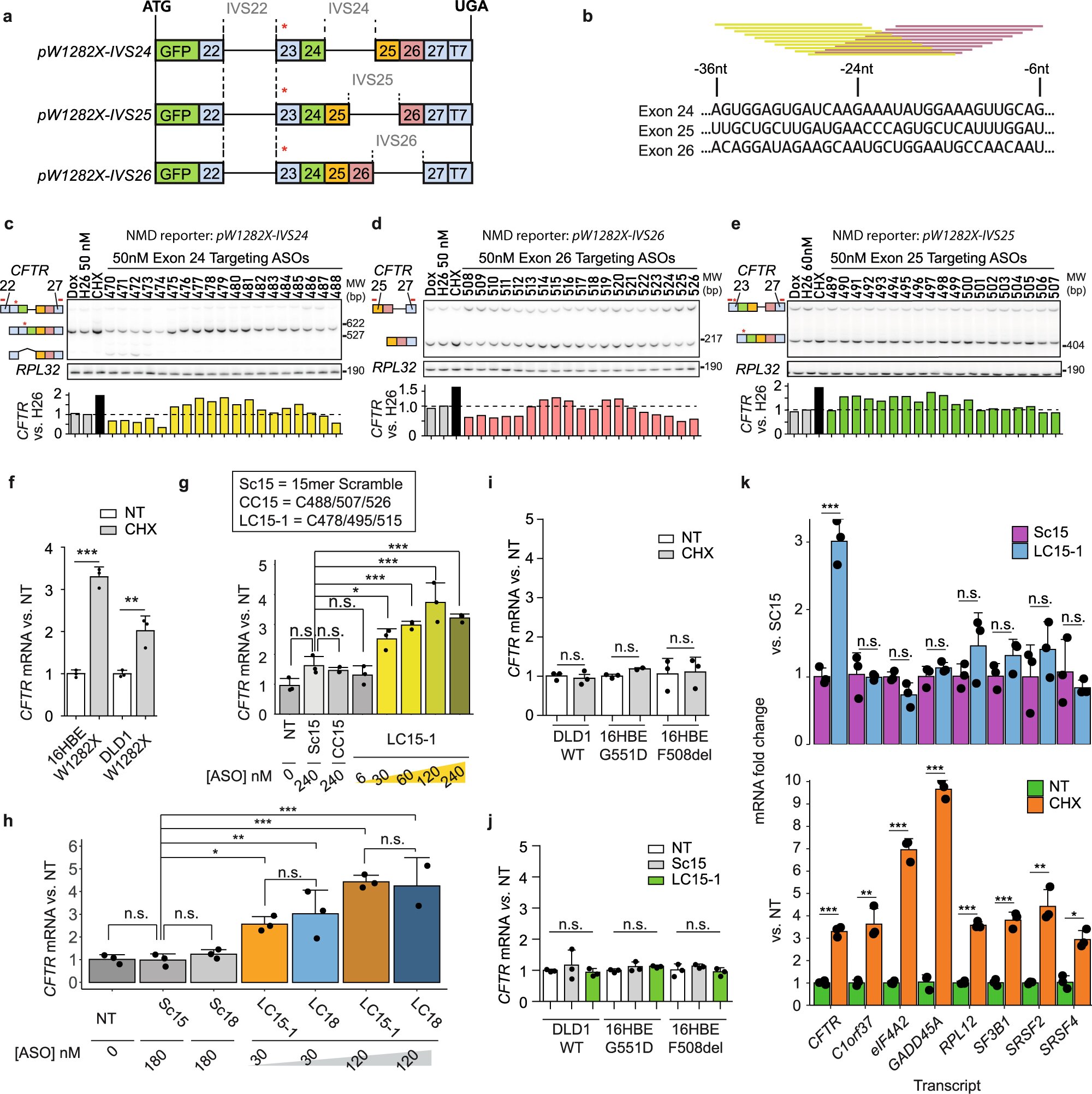
indica la ubicación de la mutación W1282X. b Esquema de la detección de ASO. Se diseñaron 19 ASO de 15 mer modificados con MOE-PS (barras amarillas y magenta) para cubrir los presuntos sitios de unión de EJC en los exones 24, 25 y 26 con una resolución de 1 nt. Las células U2OS que expresan de forma estable cada reportero NMD se transfectaron con ASO individuales dirigidos a las regiones de unión de EJC en el exón CFTR (c) 24, (d) 25, o (e) 26, respectivamente. Los niveles de ARNm informador se midieron mediante RT-PCR radiactiva, utilizando cebadores (barras rojas sobre el exón objetivo) enumerados en la Tabla complementaria 5. f Efecto de la cicloheximida en la expresión de CFTR en 16HBE -Células W1282X y DLD1-W1282X. g Efecto del cóctel LC15-1 de ASO de 15 mer sobre la expresión de CFTR en células 16HBE-W1282X. h Comparación entre los efectos de LC15-1 o LC18 en la expresión de CFTR en células 16HBE-W1282X. i Efectos de la cicloheximida sobre la expresión de CFTR en células DLD1-WT, 16HBE-G551D y 16HBE-F508del. Niveles de ARNm de j CFTR en DLD1-WT, 16HBE-G551D y 16HBE-F508del transfectados con Sc15 o LC15-1 a una concentración total nominal de 120 nM. k Niveles endógenos de ARNm sensible a NMD en células 16HBE-W1282X tratadas con cicloheximida, Sc15 120 nM o LC15-1. Todos los niveles de ARNm en (f)–(k) se midieron mediante RT-qPCR; Los niveles de ARNm de CFTR se midieron utilizando cebadores directos e inversos dirigidos al exón 22 y al exón 23, respectivamente. RPL32 sirvió como referencia interna para todos los paneles excepto h, en el que HPRT sirvió como referencia interna. NT = Sin tratamiento; Dox: doxiciclina 1 μg/mL; Sc15/18 = 15/18-mer Scramble ASO; CC15 = cóctel ASO C488/C507/C526; LC15-1=cóctel ASO C478/C495/C515; LC18 = cóctel de ASO de 18 mer C24/25/26; CHX = 1 h de incubación con 100 μg/mL de cicloheximida. Los datos se representan como valores medios ± SD. Todos los puntos de datos representan réplicas biológicas independientes. c–e (n = 1). f–k (n = 3 para todos los tratamientos, excepto n = 2 en LC18-mer 120 nM en h). Para todas las pruebas estadísticas, ns P > 0,05, *P < 0,05, **P < 0.01, ***P < 0.001. f, h (LC15-1 frente a LC18), i, k t de Student de dos colas -prueba. g, h ANOVA unidireccional con post-test de Dunnett, frente a Sc15. j ANOVA unidireccional. Crédito: Nature Communications (2022). DOI: 10.1038/s41467-022-30668-y Identificación de ASO inhibidores de NMD y evaluación de su especificidad. a Esquema de los reporteros NMD. Los números muestran la CFTR exones en los reporteros NMD. el asterisco rojo indica la ubicación de la mutación W1282X. b Esquema de la detección de ASO. Se diseñaron 19 ASO de 15 mer modificados con MOE-PS (barras amarillas y magenta) para cubrir los presuntos sitios de unión de EJC en los exones 24, 25 y 26 con una resolución de 1 nt. Las células U2OS que expresan de manera estable cada reportero NMD se transfectaron con ASO individuales dirigidos a las regiones de unión a EJC enCFTRexón (C) 24, (d) 25, o ( mi ) 26, respectivamente. Los niveles de ARNm informador se midieron mediante RT-PCR radiactiva, utilizando cebadores (barras rojas sobre el exón objetivo) enumerados en la Tabla complementaria 5. F Efecto de la cicloheximida en CFTR expresión en células 16HBE-W1282X y DLD1-W1282X. gramo Efecto del cóctel ASO de 15 mer LC15-1 en CFTR expresión en células 16HBE-W1282X. h Comparación entre los efectos de LC15-1 o LC18 en CFTR expresión en células 16HBE-W1282X. i Efectos de la cicloheximida en CFTR expresión en células DLD1-WT, 16HBE-G551D y 16HBE-F508del. j CFTR Niveles de ARNm en DLD1-WT, 16HBE-G551D y 16HBE-F508del transfectados con Sc15 o LC15-1 a una concentración total nominal de 120 nM.kNiveles endógenos de ARNm sensible a NMD en células 16HBE-W1282X tratadas con cicloheximida, Sc15 120 nM o LC15-1. Todos los niveles de ARNm en (F)–( k ) fueron medidos por RT-qPCR; CFTR Los niveles de ARNm se midieron utilizando cebadores directos e inversos dirigidos al exón 22 y al exón 23, respectivamente. RPL32sirvió como referencia interna para todos los paneles excepto h en el cual HPRTsirvió como referencia interna. NT = Sin tratamiento; Dox: doxiciclina 1 μg/mL; Sc15/18 = 15/18-mer Scramble ASO; CC15 = cóctel ASO C488/C507/C526; LC15-1=cóctel ASO C478/C495/C515; LC18 = cóctel de ASO de 18 mer C24/25/26; CHX = 1 h de incubación con 100 μg/mL de cicloheximida. Los datos se representan como valores medios ± SD. Todos los puntos de datos representan réplicas biológicas independientes.C –mi( norte= 1).F –k ( norte= 3 para todos los tratamientos, excepto norte= 2 en LC18-mer 120 nM en h ). Para todas las pruebas estadísticas, nsPAGS> 0.05, *PAGS< 0,05, **PAGS< 0,01, *** PAGS< 0,001. F , h(LC15-1 frente a LC18), i , kestudiante de dos colas t-prueba. gramo , h ANOVA unidireccional con postest de Dunnett, versus Sc15. jANOVA de una vía. Crédito:
Comunicaciones de la naturaleza
(2022). DOI: 10.1038/s41467-022-30668-y
Los oligonucleótidos antisentido, o ASO, son moléculas que se pueden usar para controlar los niveles de proteína en las células. El profesor del Laboratorio Cold Spring Harbor, Adrian Krainer, aprovechó la tecnología ASO para desarrollar el primer tratamiento aprobado por la FDA para la atrofia muscular espinal llamado Spinraza. El medicamento ha ayudado a más de 11,000 pacientes a producir más de una proteína que necesitan ciertas neuronas en la columna vertebral.
Desde entonces, Krainer ha estado buscando más formas en que los ASO puedan ayudar a tratar otros trastornos. Se ha centrado en la fibrosis quística (FQ), en la que los pacientes no producen suficiente cantidad de una proteína llamada CFTR. Su equipo descubrió cómo usar ASO para hacer más de una versión imperfecta pero aún funcional de CFTR. El descubrimiento sienta las bases para un nuevo enfoque terapéutico que puede ayudar a reducir los síntomas de la FQ y mejorar la calidad de vida de los pacientes. La proteína CFTR imperfecta es el resultado de una mutación genética. Hace que las células reciban instrucciones incorrectas para producir la proteína. Las instrucciones defectuosas se eliminan y la proteína no se fabrica, ya que, en general, las proteínas imperfectas pueden ser disruptivas. Los ASO de Krainer engañan a las células para que sigan las instrucciones defectuosas y produzcan la proteína CFTR imperfecta. Su equipo descubrió que, en este caso de FQ, tener una versión imperfecta de la proteína es mejor que no tener ninguna. Su método mejoró la función de las células pulmonares, lo que sugiere que la estrategia ASO podría mejorar los síntomas en pacientes con FQ con esta mutación.Publicado en
Comunicaciones de la naturaleza
Edición de ARN para solucionar problemas de proteínas en la fibrosis quística Más información:Young Jin Kim et al, Orientación de la descomposición del ARNm mediada por tonterías específicas de genes para la terapia de la fibrosis quística, Comunicaciones de la naturaleza
DOI: 10.1038/s41467-022-30668-yProporcionado por el Laboratorio de Cold Spring Harbor
Citación
: Un nuevo enfoque de tratamiento para la fibrosis quística (14 de julio de 2022) recuperado el 14 de julio de 2022 de https://medicalxpress.com/news/2022-07-treatment-approach-cystic-fibrosis.html Este documento está sujeto a derechos de autor. Aparte de cualquier trato justo con fines de estudio o investigación privados, ninguna parte puede reproducirse sin el permiso por escrito. El contenido se proporciona únicamente con fines informativos.
Fuente